本文經訊聯基因數位股份有限公司同意轉載
關於IPA 與IPA Interpret
QIAGEN IPA 是一款雲端生物資訊分析軟體,可用來分析以下多種實驗數據:
- RNA-seq 表現量資料
- 基因晶片(microarray)
- SNP 陣列
- 蛋白質體學
- 代謝體學
- 其他產生基因清單的小型實驗(如PCR)
除了資料分析,IPA 還能搜尋特定基因、蛋白質、化學物質、疾病與藥物的資訊,建立專屬的生物模型。透過 IPA 的分析和建模功能,再搭配結構化整理的生物與化學研究發現資料庫,能幫助您更加了解研究數據在更大的生物或化學系統中的意義。
IPA Interpret 為IPA 中包含的元件,可透過快速且現代化的網頁介面呈現 IPA Core Analysis 的結果,並與他人分享。
全新的 IPA Interpret 主頁中整合您的分析連結
您可以快速地在新增的 IPA Interpret 主頁中找到您所有可存取的分析報告(包含您與他人的共用分析報告)並開啟。分析報告可依以下屬性進行篩選:
- 專案名稱
- 日期
- 分析名稱
甚至可以根據IPA 桌面版無法使用的屬性進行篩選,例如:
- 分析的基因數量
- 使用的參考集合(reference set)
- 分析的擁有者(在共享分析中特別實用)
分析預設會依照時間由新到舊排列,讓您先看到最新的分析結果。
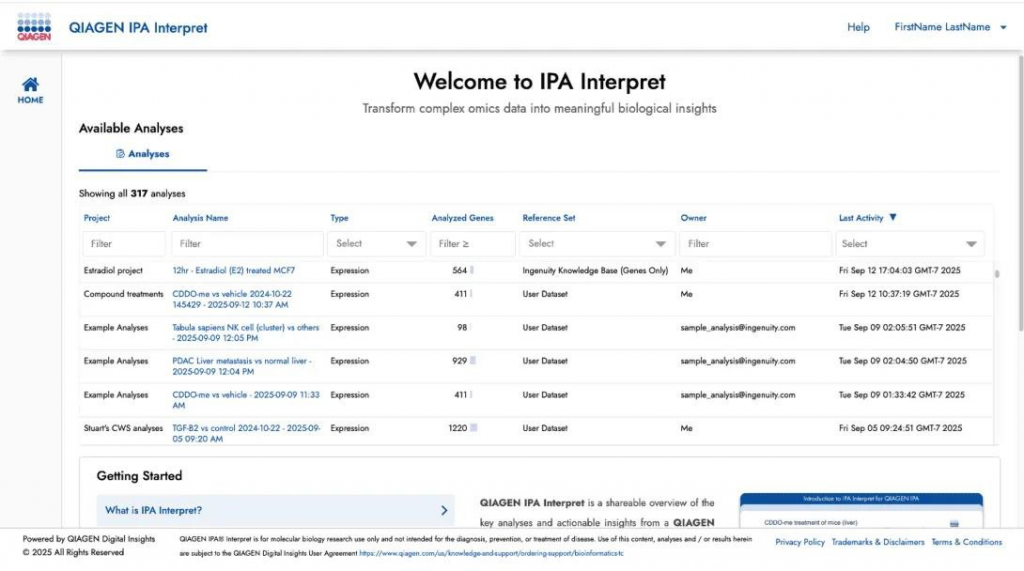
您的主頁上所看見的分析列表可能會與此螢幕截圖不同
但所有使用者都會看到一小組隨附的範例分析
請注意,僅 2024 年 9 月之後執行的分析結果會顯示於此列表中。
此外,儲存在協作工作區(Collaboration Workspaces, CWS)中的分析,也會自動顯示在此處。此功能適用於擁有機構版或商業版授權(concurrent licenses)的使用者,內部生物資訊團隊可透過此機制將分析結果直接發佈給最終使用者。
如需更多資訊,請聯絡訊聯基因數位 巨量生物資料工程部
主頁的下方區域包含「快速入門(Getting Started)」區塊,其中提供說明影片與協助連結(見圖二)。這些資源將對於與您共用分析的非 IPA 使用者特別有幫助。
觀看影片和點擊連結來學習更多
若要從IPA Interpret 報告頁面返回主頁,請點擊登入連結(未登入者需登入),然後點擊報告頁面右上角的主頁圖示(圖三)

點擊主頁圖示(紅框標示)以返回主頁頁面
IPA Interpret 資料集火山圖解讀
在IPA Interpret 以上傳資料集所繪製的火山圖(volcano plot)中,您可以透過標示圖中特定的基因或蛋白質群集來進一步探索這些分子。此項全新的功能將有助於您聚焦資料中您認為最重要的部分。
例如,您可以標示出表現變化最顯著、且為細胞外蛋白(extracellular proteins)的基因。
您可以透過在資料集表格中同時使用 Location 篩選器與 Fold Change 篩選器(見圖四紅框標示)來達成此目的。
此功能會淡化不符合篩選條件的基因,以突顯您感興趣的目標。
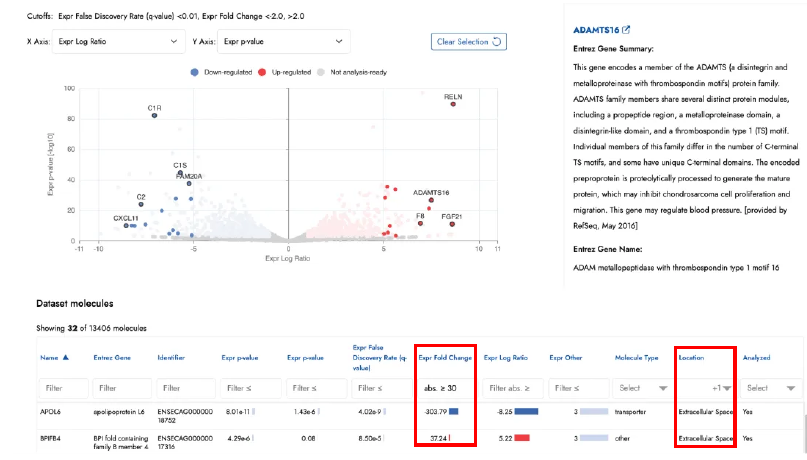
資料集表格已篩選出Fold Change 絕對值 ≧ 30 且 Location 為「細胞外空間(Extracellular Space)」的基因
然後點擊圖中某些點,即可在這些點上顯示對應的基因名稱標籤
如果您想要查看描述中含有「myosin(肌球蛋白)」的基因的分布概況,可以在 Entrez Gene 欄位篩選器中輸入該關鍵字來進行篩選(見圖五紅框標示)。
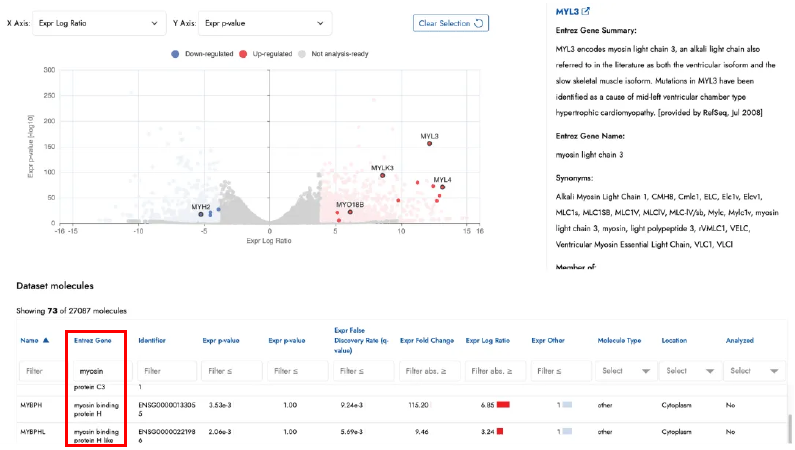
資料集表格已篩選出Entrez Gene 描述中包含「myosin」的基因
然後點擊圖中的某些點,即可在這些點上顯示對應的基因名稱標籤
其他 IPA Interpret 的精進與錯誤修正之處
- 在分析頁面的標題列中新增了說明連結(Help link)
- 在分析主頁上方新增了「如何分享(How to share)」的彈出式視窗
- 修正了兩個名稱中包含「+」符號的Canonical Pathways(經典路徑)的問題
- 已改善泡泡圖(bubble chart)中過長路徑名稱被截斷的問題
- 已修正長條圖(bar chart)中「排序依據(Sort by)」功能的問題
IPA 桌面版具更快的分析速度
使用預設的生物篩選條件(biological filters)進行分析的執行時間較先前版本短。這是因為以下「子分析(sub-analyses)」在使用預設篩選時的執行速度已大幅提升:
- Canonical Pathways
- Upstream Regulators
- Diseases and Functions
- Tox Functions
然而,無論篩選條件如何,下列子分析項目仍會以過往相同的速度執行:
- Graphical Summary
- Regulator Effects
- ML Disease Pathways
- Tox Lists
- Causal Networks(可選)
- Interaction Networks(可選)
- My Pathways / My Lists(可選)
「生物篩選條件(Biological filters)」是指分析設定中的項目,例如「物種(Species)」或「組織與細胞株(Tissues & Cell Lines)」,如圖六所示。
例如,若您將分析的物種設定從預設的「All」改為「mouse(小鼠)」,該分析將會以較慢的速度執行。
而若您使用預設的生物篩選條件,並排除前述的選用子分析(optional analyses),分析速度便可提升至最多五倍。至於其他設定(如參考集合或閾值)則可自由調整,不會影響上述子分析的執行速度。
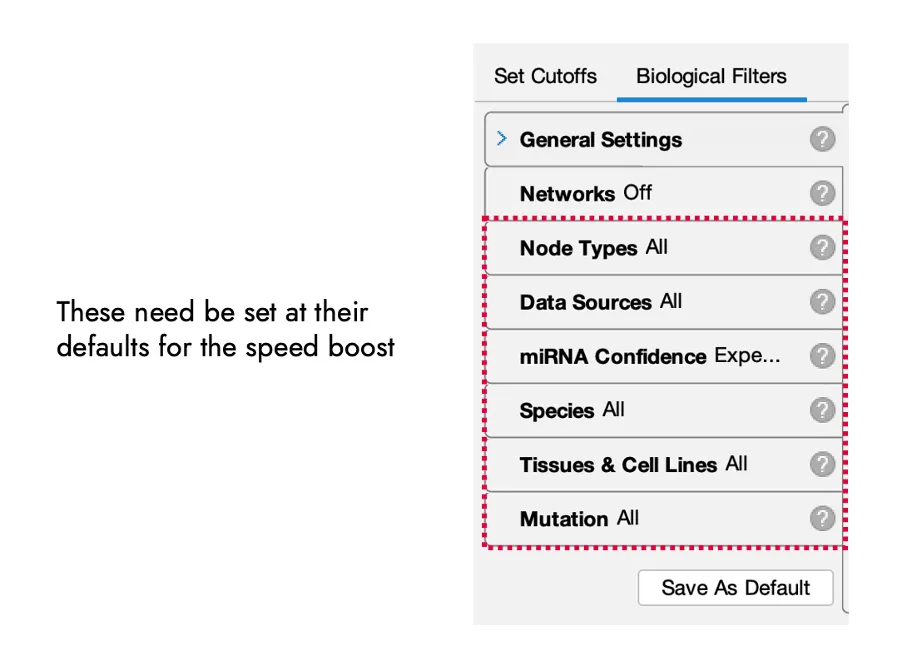
這些篩選條件的預設值皆為 「 All 」 ,唯獨 miRNAConfidence miRNA 可信度) 篩選條件應設定為 「 Experimentally Observed (實驗驗證)」 。
如果您在 General Settings(一般設定)或 Networks(網路)中啟用了任何選用分析(optional analyses),分析執行時間將會延長,延遲程度取決於所包含的分析項目。在上述 Networks 分析中的最耗時的選用子分析為:
- Causal Networks(因果網路)
- Interaction Networks(交互作用網路)
精進 IPA 桌面版中的網路和路徑的可讀性
在IPA 桌面版中,關係連線(relationship lines)現在預設為淡化顯示,以便更清晰地查看路徑或網路中的節點。這些關聯線條(edges)過去顏色偏重,可能造成整體圖像不易辨識。圖七展示了淡化與未淡化連線之間的差異,同時也顯示了IPA 桌面版中的PathTracer 功能表,您可在此調整連線的視覺化設定。
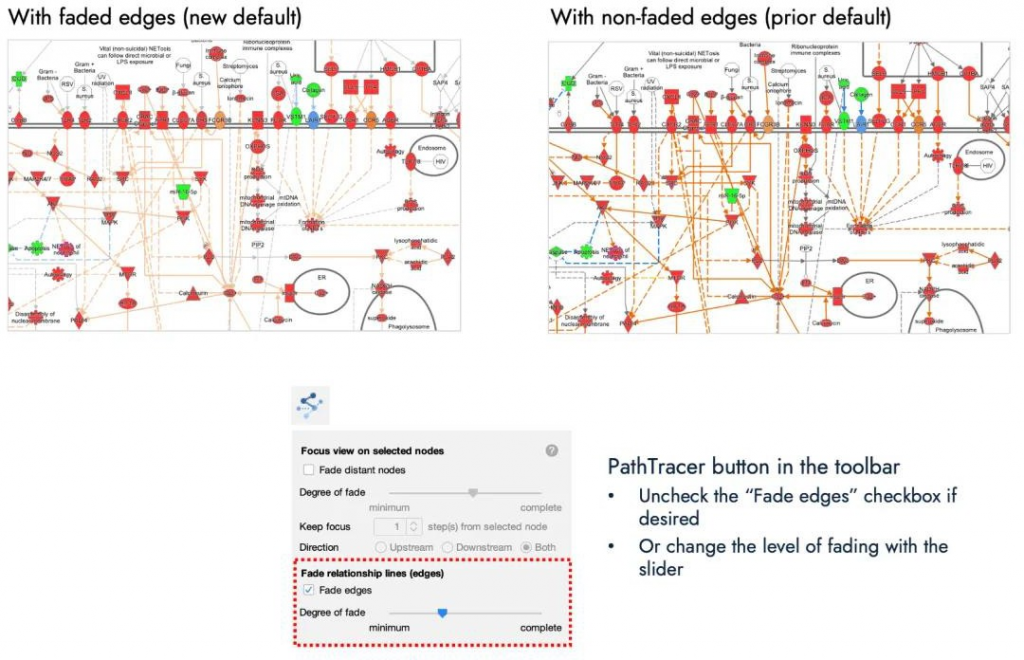
您可以透過上方所示的PathTracer 選項
自由調整連線的淡化程度,或完全關閉淡化效果
IPA 桌面版新增 OmicSoft Explorer 的連結
IPA 桌面版主視窗右上角已新增導向OmicSoft Explorer 的連結,而非先前的OmicSoft Land Explorer。
擁有Analysis Match Explorer 授權的使用者,可點擊此連結,在全新的、易使用的網頁介面中搜尋並探索OmicSoft 的大型精選實驗資料庫。

擁有Analysis Match 授權的使用者可以點擊紅色虛框內的連結探索OmicSoft Explorer 龐大的精選實驗資料集
您可以造訪此網頁,以進一步了解如何存取與探索這個支援IPA「Analysis Match」功能的獨特資料來源。
內容更新
新增六條 Ingenuity 訊號傳導路徑:
- 精卵融合訊號傳導路徑(Egg-Sperm Fusion Signaling Pathway)
- 溶體定位訊號傳導路徑(Lysosome Positioning Signaling Pathway)
- 心肌素訊號傳導路徑(Myocardin Signaling Pathway)
- 淋巴細胞活化訊號分子家族(SLAMF 之訊號傳導路徑)(SLAMF Signaling Pathway)
- 硫氧還蛋白訊號傳導路徑(Thioredoxin Signaling Pathway)
- 維生素K 訊號傳導路徑(Vitamin K Signaling Pathway)
新增 29 條 Reactome 路徑:
- BMAL1:CLOCK, NPAS2 活化晝夜節律表達 (BMAL:CLOCK, NPAS2 activates circadian expression)
- 染色質修飾酶 (Chromatin modifying enzymes)
- 環丙沙星的吸收、分佈、代謝和排泄 (Ciprofloxacin ADME)
- B 和T 淋巴球衰減因子(BTLA)共抑制 (Co-inhibition by BTLA)
- 細胞毒性T 淋巴細胞抗原-4(CTLA4)共抑制 (Co-inhibition by CTLA4)
- 程序性死亡蛋白-1(PD-1)介導之共抑制 (Co-inhibition by PD-1)
- 誘導型T 細胞共同刺激分子(ICOS)介導之共刺激 (Co-stimulation by ICOS)
- CRY 和 PER 蛋白的降解 (Degradation of CRY and PER proteins)
- MLL3 和MLL4 複合體對基因表達的表觀遺傳調控 (Epigenetic regulation of gene expression by MLL3 and MLL4 complexes)
- BMAL(ARNTL)、CLOCK 和 NPAS2 的表達 (Expression of BMAL (ARNTL), CLOCK, and NPAS2)
- 肌萎縮蛋白-醣蛋白複合體形成 (Formation of the dystrophin-glycoprotein complex (DGC))
- 鞘糖脂運輸 (Glycosphingolipid transport)
- 高層流剪應力活化內皮細胞中 PIEZO1 和 PECAM1:CDH5:KDR 的訊號傳導 (High laminar flow shear stress activates signaling by PIEZO1 and PECAM1:CDH5:KDR in endothelial cells)
- 整合素至細胞骨架的訊號傳導途徑 (Integrin to Cytoskeleton Signaling Pathway)
- 機械負荷活化骨細胞中 PIEZO1 和整合素的訊號傳導 (Mechanical load activates signaling by PIEZO1 and integrins in osteocytes)
- 粒線體未摺疊蛋白反應 (Mitochondrial unfolded protein response (UPRmt))
- IFN 刺激基因對宿主反應的調控 (Modulation of host responses by IFN-stimulated genes)
- NIK →非典型 NF-kB 訊號傳導 (NIK → noncanonical NF-kB signaling)
- 細胞核 RNA 降解 (Nuclear RNA decay)
- 磷酸化 BMAL1:CLOCK 活化核心時鐘基因的表達 Phosphorylated BMAL1:CLOCK activates expression of core clock genes)
- BMAL1 和CLOCK 的磷酸化和核轉位 (Phosphorylation and nuclear translocation of BMAL1 and CLOCK)
- CRY:PER:激酶複合體的磷酸化和核轉位 (Phosphorylation and nuclear translocation of the CRY:PER:kinase complex)
- CLOCK 磷酸化與BMAL1 在標靶基因啟動子區域的乙醯化 (Phosphorylation of CLOCK, acetylation of BMAL1 at target gene promoters)
- 蛋白酶體組裝 (Proteasome assembly)
- RORA, B, C 和 NR1D1 (REV-ERBA)調節基因表達 (RORA, B, C and NR1D1 regulated gene expression)
- SWI/SNF 染色質重塑複合體 (SWI/SNF chromatin remodelers)
- CRY:PER 激酶複合體抑制 BMAL:CLOCK 複合體的反式活化 (The CRY: PER: kinase complex represses transactivation by the BMAL:CLOCK complex)
- 紊流(震盪、擾亂)剪應力活化內皮細胞中 PIEZO1 和整合素的訊號傳導 (Turbulent flow shear stress activated signaling by PIEZO1 and integrins in endothelial cells)
- WNT/Ca2+訊號傳導途徑 (WNT/Ca2+ Signaling Pathway)
重新命名的Reactome 路徑
| 舊命名 | 新命名 |
| Arachidonate acid metabolism | Arachidonate metabolism (花生四烯酸代謝) |
| Synthesis of Lipoxins (LX) | Biosynthesis of Lipoxins (LX) (脂氧素的生物合成) |
| Carnitine metabolism | Carnitine shuttle (肉鹼穿梭) |
| Costimulation by the CD28 family | Co-stimulation by CD28 (由CD28 介導的共刺激) |
| Serine biosynthesis | Serine metabolism (絲氨酸代謝) |
| Vitamin E | Vitamin E transport (維生素K 轉運) |
刪除的Reactome 路徑
| 刪除的路徑 | 刪除理由 |
| Circadian Clock (晝夜節律時鐘) | 為多條路徑的上層路徑 |
| Chromatin Organization (染色質構建) | 為多條路徑的上層路徑 |
| NR1D1 (REV-ERBA) represses gene expression (NR1D1 抑制基因表達) | 合併成名為「RORA,B,C 和NR1D1 (REV-ERBA) 調節基因表達」的路徑 |
| RORA activates gene expression (RORA 活化基因表達) |
知識庫更新
新增的 12 萬 3 千多筆新發現,使知識庫總數達到 1,470 萬筆。
- 超過 79,000 筆來自專家文獻註解
- 超過 11,000 筆BioGrid 蛋白質交互作用資料
- 超過 67,000 筆ClinVar 癌症突變資料
- 超過 1,400 筆ClinicalTrials.gov 藥物與疾病資料
- 超過 1,400 筆 ClinicalTrials.gov 靶點與疾病資料
- 超過 4,500 筆基因本體(Gene Ontology)研究發現
- 超過 1,300 筆IntAct 蛋白質交互作用資料
OmicSoft 資料集
OmicSoft 中新增了 10,159 個資料集,使總資料集數達到 266,592。
| Land | Repository | Q2 | Q3 | Increase |
| DiseaseLand | HumanDisease | 39,650 | 40,155 | 505 |
| MouseDisease | 29,564 | 30,234 | 670 | |
| RatDisease | 10,269 | 10,269 | ||
| LINCS | 25,880 | 25,880 | ||
| OncoLand | OncoHuman | 25,472 | 26,533 | 791 |
| OncoMouse | 1,516 | 1,516 | ||
| TCGA | 4,854 | 4,854 | ||
| ENCODE RNA Binding | 486 | 486 | ||
| ClinicalProteomic Tumor | 3090 | 3419 | 329 | |
| NCI Patient-Derived Models | 552 | 552 | ||
| Single Cell Land | SingleCellHuman | 194 | 194 | |
| SingleCellHumanUmi | 89,283 | 88,525 | -758* | |
| SingleCellHumanHCL | 1476 | 1476 | ||
| SingleCellMouse | 81 | 81 | ||
| SingleCellMouseUmi | 23,242 | 29,481 | 6,239 | |
| Normal Cells and Tissues | Human Tissues (GTEx) | 1,312 | 2937 | 1,625 |
原始文章來源:https://digitalinsights.qiagen.com/products/qiagen-ipa/latest-improvements/current-line/