範例:進行C. eleganSUMO-conjugating enzyme UBC9 yeast two-hybrid實驗後,發現有一個蛋白質會與UBC9有交互作用,長度有702胺基酸,進行蛋白質序列分析。
1. 可將你的蛋白質序列送到一些常見的工具進行搜尋,例如NCBI的 BLAST(Basic Local Alignment Search Tool)中的blastp程式進行比對(網址:http://www.ncbi.nlm.nih.gov/BLAST/)
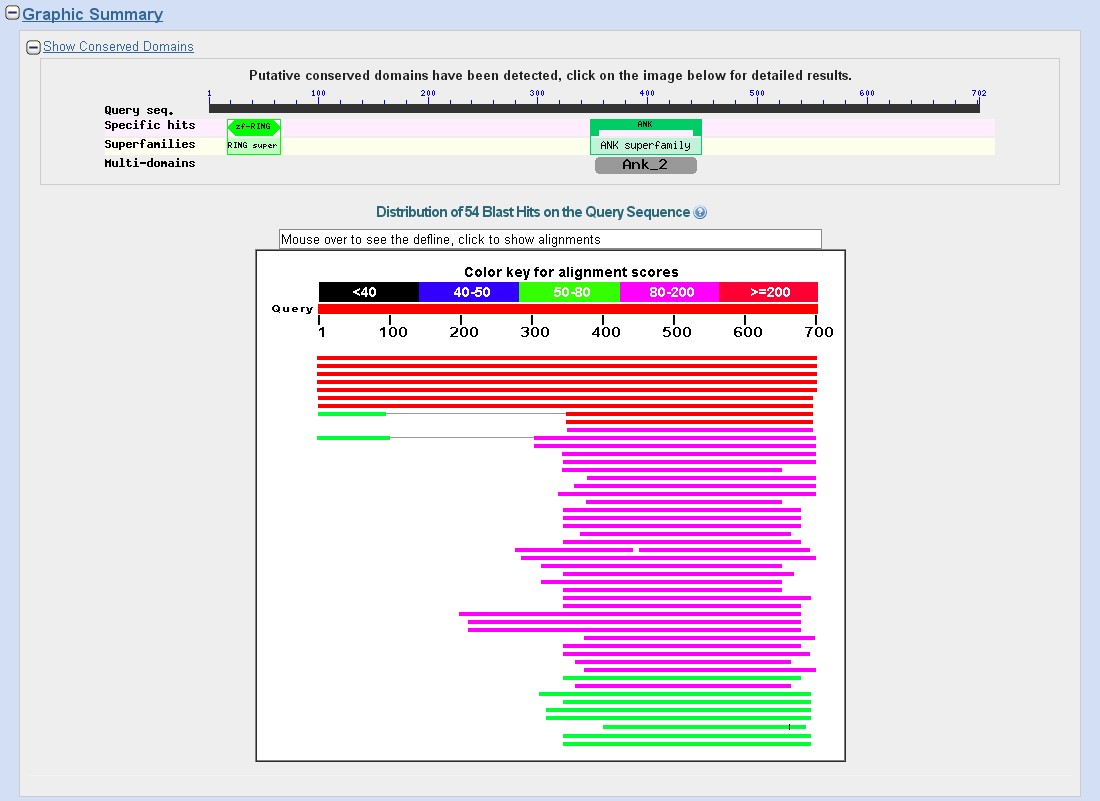
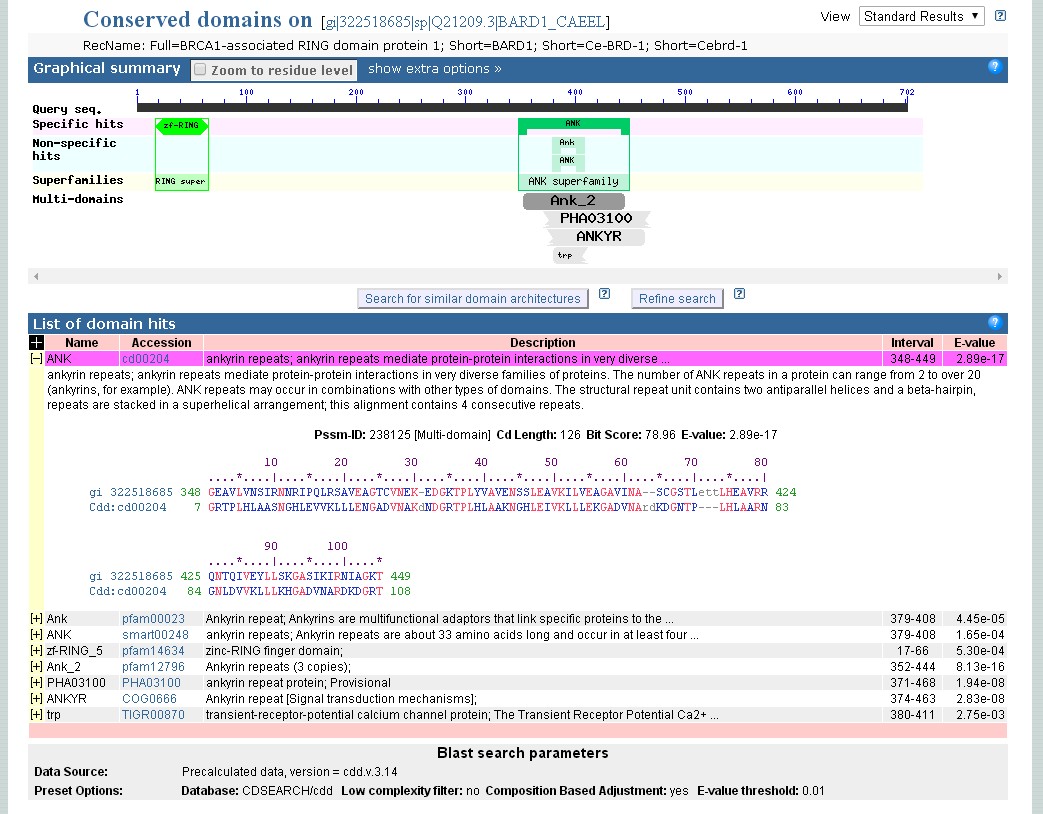
搜尋結果顯示此一蛋白質是C. eleganBRCA1-associated RING domain protein 1 (brd-1),同時也知道此一蛋白質包含Zinc-Ring finger 和Ankyrin repeats的序列特性。
序列的motifs 或domains 也可從NCBI 的 CD-Search (http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi) 搜尋得知。CD-search 是一個用來搜尋CDD的介面,CDD包含Pfam, SMART, COGs 和 MMDB 輸入的具有保留的蛋白質patterns或motifs。 利用事先計算的位置相關的計分矩陣與搜尋的序列進行比對,搜尋的結果顯示為一對一的排比和可連結的以不同顏色表示的domain區塊。 展開”+”即可顯示序列相似的程度。
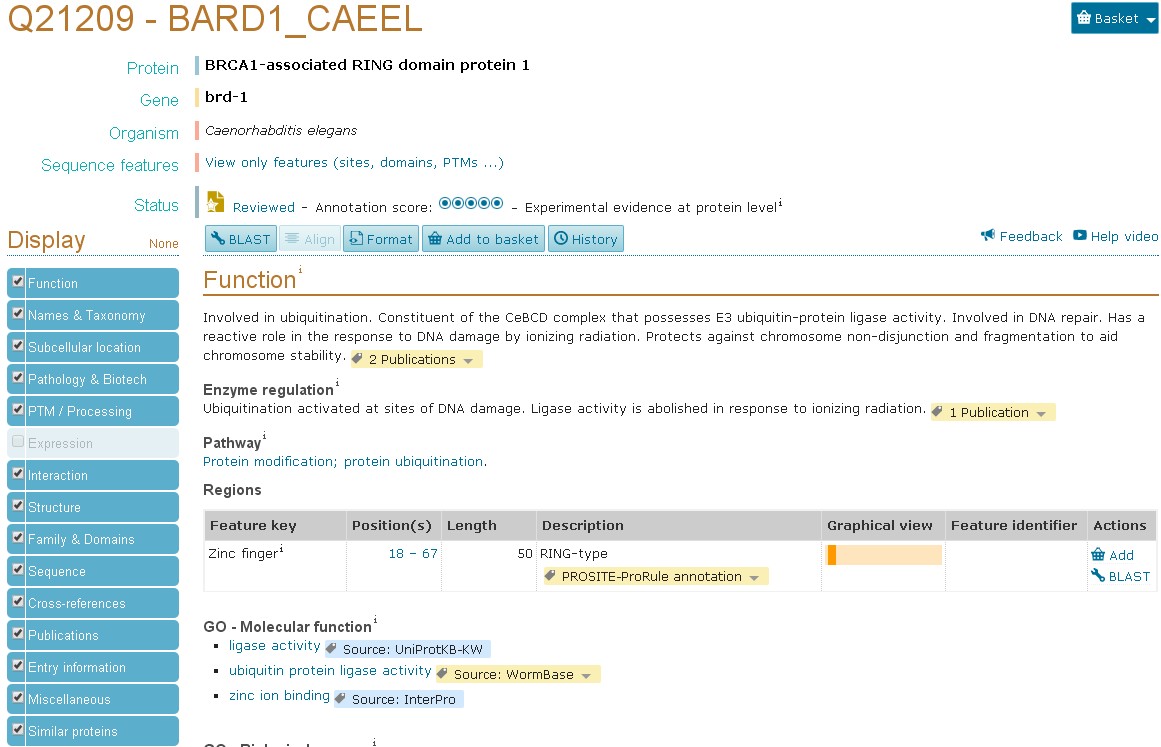
2. 將你的蛋白質序列送到UniProt 進行BLAST搜尋 (http://www.uniprot.org/blast/)
找到完成相同的C. eleganBRCA1-associated RING domain protein 1 (Q21209)後,會得到更多與brd-1相關的訊息。
以上內容感謝生醫所孫慶姝小姐提供,希望對您們有幫助。