Purpose: 在蛋白質序列進行大規模的 (large scale) 搜尋潛在的基質並以模擬三維結構 (model of 3D structure)顯示。
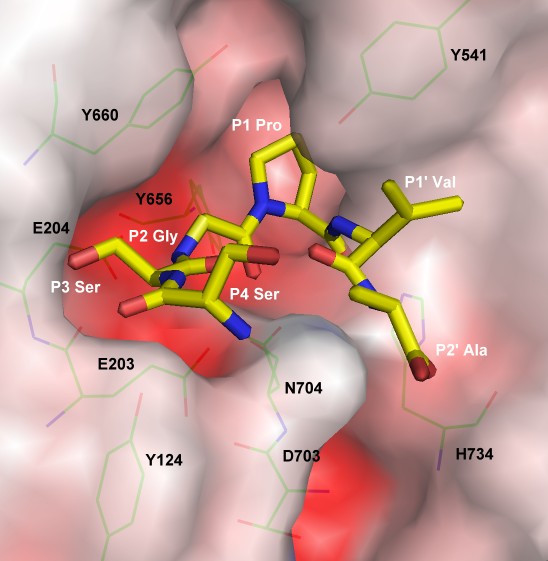
1. 根據文獻資料顯示,成纖維細胞活化蛋白的基質具有蛋白質序列專一性 (sequence specificity) (FEBS lett, 2006, 580:1581-1586),基質的P1位置為脯氨酸 (Proline) 和 P2位置為甘氨酸 (Glycine).
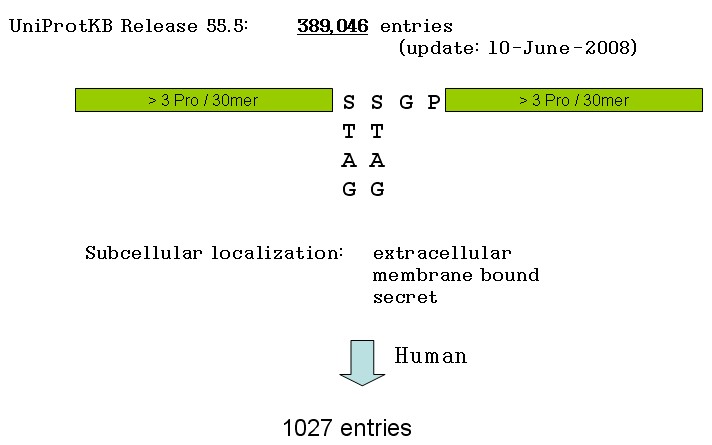
2. 從蛋白質序列資料庫UniProtKB (http://www.uniprot.org/)下載蛋白質序列,序列之間是以“//”區隔。
3. 設定名稱“human” 、“extracellular”、“membrane bound”、“ secret”。從中搜尋設定名稱,再將選擇符合條件的序列進行以下字串搜尋。
4. 符合字串搜尋並經過蛋白質三級結構分析或二級結構預測,係位於結構彎曲部分 (loop)的個案則為接下來實驗的對象。其中 SPRY2 (Protein sprouty homolog 2)被成纖維細胞活化蛋白所裂解 (cleavage)。
5. 從蛋白質結構資料庫 (PDB; http://www.rcsb.org/pdb/home/home.do)下載成纖維細胞活化蛋白的三級結構 (PDB code 1Z68 and 1NU8),運用InsightII軟體 [InsightII 為舊版軟體,現在使用的是BIOVIA Discovery Studio (http://accelrys.com/products/collaborative-science/biovia-discovery-studio/)] 以diprotin A的結構為基礎,產生SPRY2的SSGPVA結構片段,將此一結構片段對接 (manually docked) 至成纖維細胞活化蛋白的活性位置,如下圖。
6. 這些結果發表在
Cleavage-site specificity of prolyl endopeptidase FAP investigated with a full-length protein substrate. Chih-Hsiang Huang, et al. J. Biochem. 2011 49: 685-692. [PubMed link]
※以上所描述的過程僅僅是表達生物資訊資料庫及工具的應用及對實驗的輔助,事實上,實驗的過程是最重要也是最主要的貢獻者。若有誤導(misleading)或得罪(offensive)之處,敬請見諒並給予賜教,謝謝。
以上內容感謝生醫所孫慶姝小姐提供,希望對您們有幫助。