Cn3D是一個運用視覺化來操作觀看生物分子序列、三維結構、成對或多個疊加的三維結構及其相對應的序列排列的工具。在Cn3D網頁中選擇 INSTALL (https://www.ncbi.nlm.nih.gov/Structure/CN3D/cn3d.shtml)依照你的硬體操作平台(Windows或Macintosh)來下載及安裝獨立版本 (standalone version)的Cn3D。開啟後即出現一個以「開放式圖形庫」(openGL)為主具有高畫質的分子結構視窗(structure window),其中File的選項控制檔案的輸入和輸出。此處所使用的三維結構案例4GED已經下載為cn3的檔案,所以Open即可開啟4GED的結構,但是Cn3D無法直接讀進pdb格式檔案,必須經由Network Load選項及填入PDB或MMDB代碼來取得。
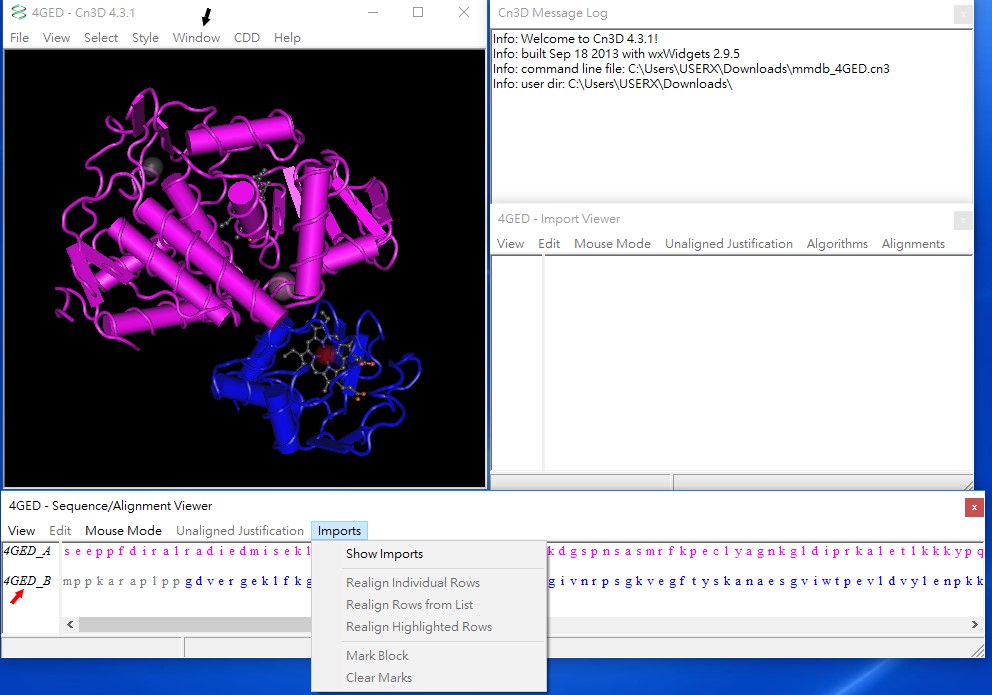
當4GED的結構開啟後,分子結構視窗會出現4GED的三維結構,另外序列視窗出現此一結構中兩個分子的序列。Window項目下除了序列視窗還有訊息視窗(Message log),可以用來追蹤操作過程。序列視窗項目中Imports的Show Imports提供多項的應用功能,例如Algorithms和Alignments。按壓序列視窗分子代碼4GED_A或4GED_B,會自動透過網路瀏覽器連接至NCBI蛋白質資料庫。選擇View→Find Pattern並填入想檢視的殘基片段,如AHT,其中His192已知是鐵離子結合殘基,按壓Ok後,此一片段在序列中顯現黃色背景,同時在結構中也會以黃色來顯示出此片段。
首先,結構視窗的基本操作為按壓滑鼠左鍵並拖動滑鼠來轉動蛋白質結構(rotation) 、Ctrl+按壓滑鼠左鍵可放大或縮小及Shift+按壓滑鼠左鍵則移位(translocation)視窗中的結構。以下的步驟用來改變結構及序列視窗的內容:
1. File → Set Fonts → Structure Window,選擇字型為Arial、字型樣式為粗斜體及大小為12,然後File → Set Fonts→ Sequence Windows,選擇字型為Consolas、字型樣式為標準及大小為12。
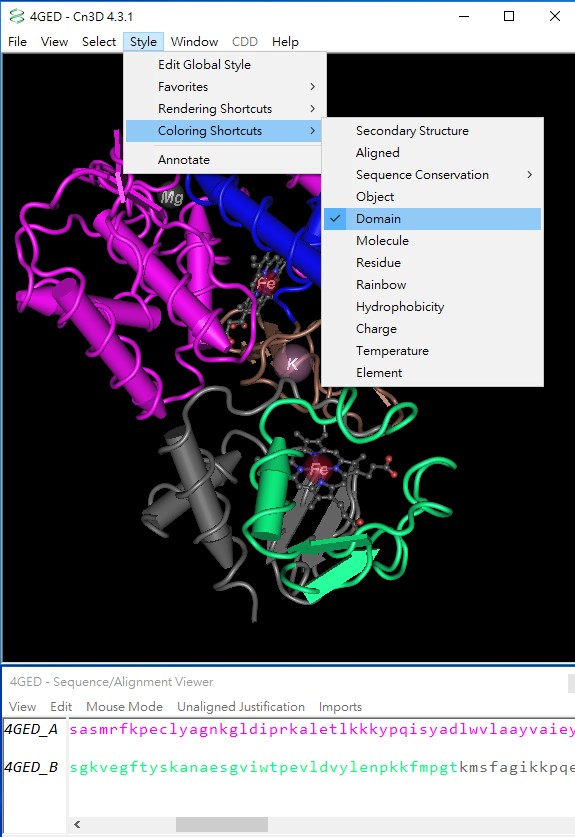
2. Style → Coloring Shortcuts → Domain後,結構顏色則顯示CDD定義的結構區域。
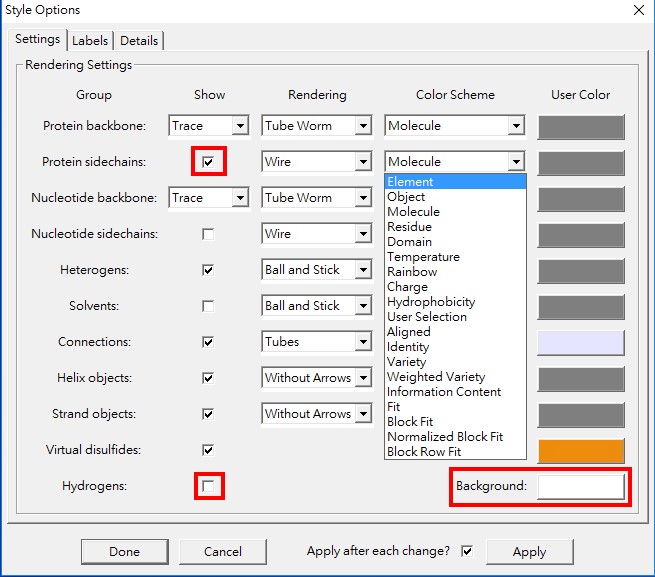
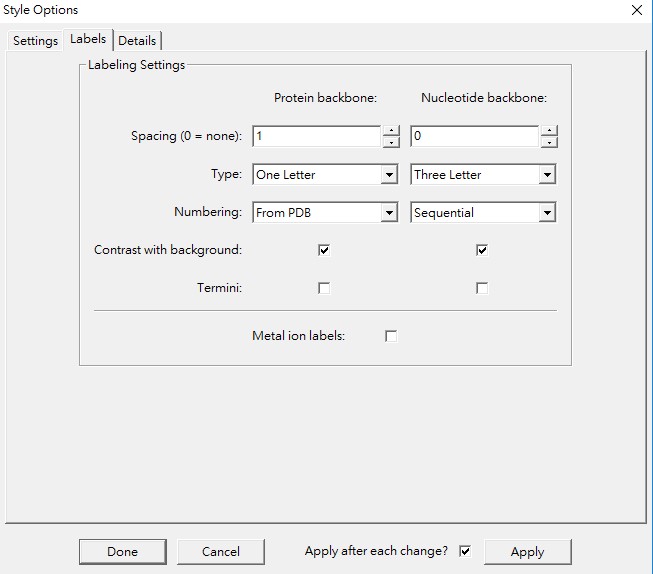
3. Style → Edit Global Style後,一個選項(Options)視窗出現,其中有三種設定項目,分別是Settings、Labels和Details。在Settings中,Helix objects和Strand objects選擇With Arrows,顯示出序列由N端到C端的次級結構。在Labels中勾選Metal ion label,結構中的金屬或化學元素被標示出來並顯示為Arial、粗斜體及大小為12的字型。在Details中,將Helix radius由原先設定的1.8改為1.5,結構視窗中的柱狀結構則隨之變更,修改後的兩個視窗如下圖。將所變動的結構及序列儲存成(Save As)另一個新的cn3格式檔案,再一次打開新檔案則會出現變動後的結構及序列,或者輸出為PNG格式(Export PNG)的圖形檔案。
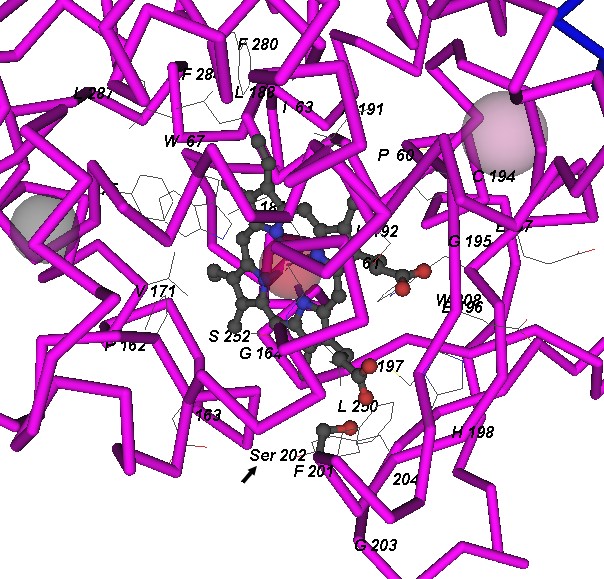
其次,取消結構的次級結構的方法為Style→Rendering Shortcuts→Tubes,按壓結構中的Heme分子並突出,選擇Select→Select by Distance來標示此分子周圍距離5Å之內與其有交互作用的殘基群。選擇Style→Annotate,在User Annotations中點擊New,並填入heme binding→Edit Style,則會出現Style Options的表單,在表單的Settings取消Hydrogens、勾選出Protein backbone、顏色選Element及Background選擇白色。在表單的Labels中Protein backbone項目的Spacing為1、Type為One Letter及Numbering為From PDB。
這些有交互作用的殘基群中,為了標示Ser202的位置,在序列中突出Ser202,選擇Style→Annotate,在User Annotations中點擊New,並填入Ser202→Edit Style,在Style Options表單的Rendering選出Ball and Stick。在表單的Labels中Protein backbone項目的Type改為Three Letter。最後,改變背景顏色則選擇Style → Edit Global Style,Background改成白色。
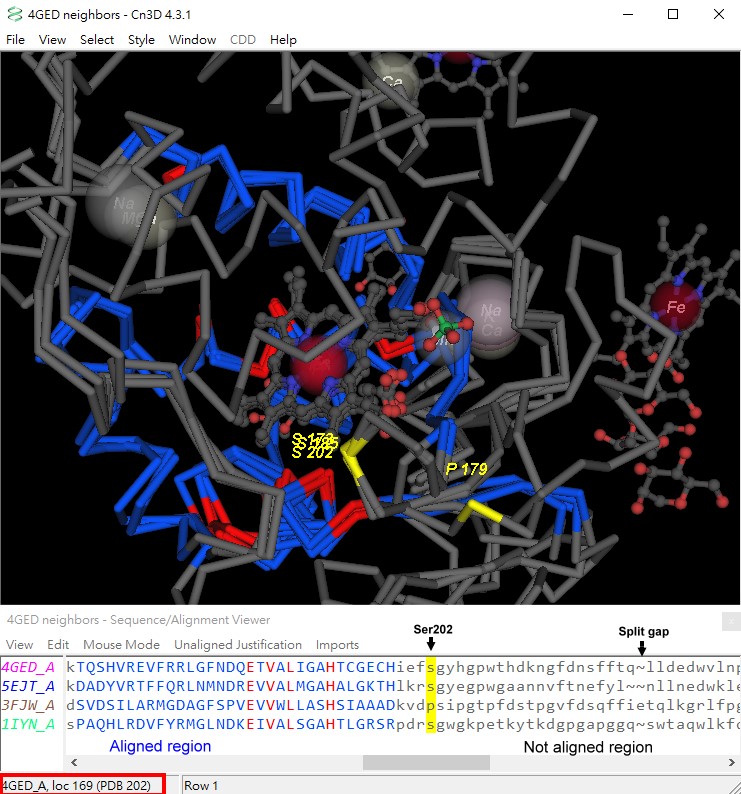
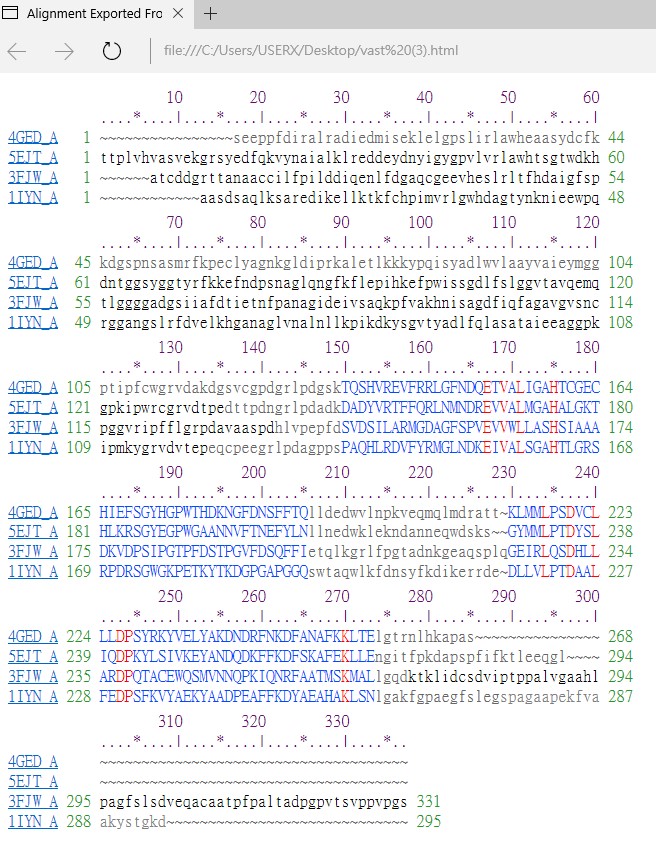
Cn3D不僅僅可以用來操作查看單一的結構,也可以作為多個經過比對之後疊加結構的顯示器。在Part I介紹了NCBI的VAST結構排列比對工具,並以4GED5中A鏈(Chain A; Ascorbate Peroxidase; APX)的domain 2 為範例,下載4GED_A與其他三個物種的結構:5EJT_A (酵母菌Yeast Ferryl Cytochrome C Peroxidase)、3FJW_A (杏鮑菇Pleurotus eryngii Versatile Peroxidase) 和1IYN_A (煙草tobacco Ascorbate Peroxidase)的疊加結構及其對應的序列排列為cn3形式的檔案,以Cn3D開啟此一檔案。此時,Cn3D序列視窗亦可作為4GED_A及其VAST找到的相似結構(也稱為鄰居)的序列排列顯示器(Sequence/Alignment Viewer),這裡的序列比對是基於結構的比對結果,與常用序列比對工具例如BLAST和Clustal系列有一些差異。結構和序列視窗中顯現藍色大寫字型的區域即為結構相似可排列的結果,其中紅色的區域是這四者序列具有一致性的胺基酸,未排列或無法排列的區域則以灰色小寫字型表示。
滑鼠游標在序列視窗移動時,會顯示該胺基酸在結構中的序列號碼,為了標示Ser202的位置及在序列中突出Ser202,在序列視窗選擇Mouse Mode →Select Columns,點擊4GED_A序列中的S202後,相對的胺基酸在四種物種則呈現黃色背景,同時結構中的位置也轉變為黃色。在結構視窗選擇Style→ Annotate,在User Annotations中點擊New,並填入Ser202→Edit Style,在Style Options表單的Labels中Protein backbone項目的Spacing為1、Type為One Letter及Numbering為From PDB。圖像中顯示Ser202於其中三種物種被保留,相對的胺基酸位於杏鮑菇(3FJW_A)的序列是Pro179,它的結構位置亦相異於其他三種物種。
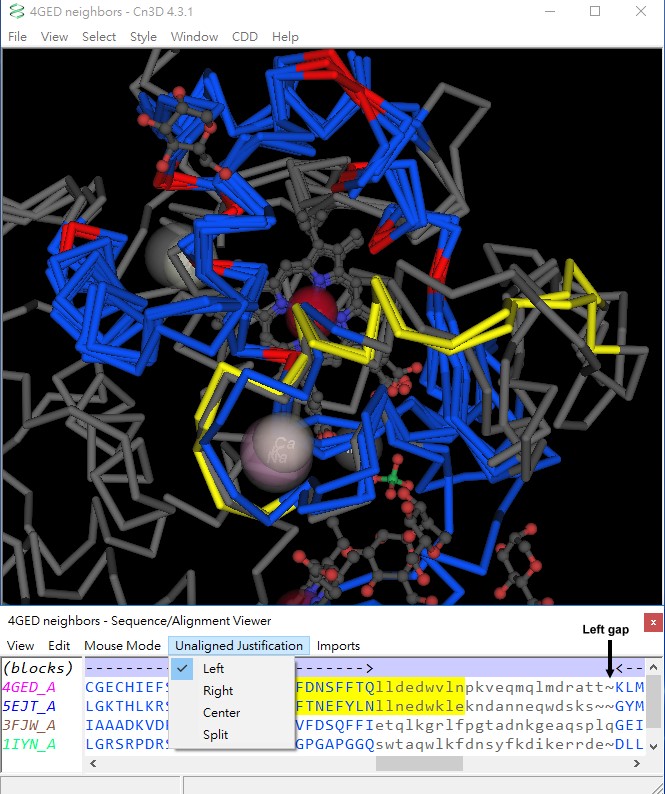
當出現結構無法比對的間隙(gap)在序列比對會以“~”來表示,原始的疊加結構及其對應的序列排列的檔案中,間隙的位置是以分裂(Split)來顯示,將間隙置放在無法比對的序列中央。因此造成4GED_A和5EJT_A序列之間排列的錯誤,修正的步驟為選擇Unaligned Justification→Left,則間隙移到右側,使得黃色背景的序列之間排列更為合理。另一方面,調整結構可排列的區域的方法為選擇 Edit→ Enable Editor,視窗中序列的上面出現淡紫色的區塊指示(Block indicator),其間以箭頭及虛線表示藍色大寫字型的區域,可按壓箭頭向左右移動來加長或縮短可排列的區域,亦即藍色大寫字型的區域。
調整後的序列排列可經由View→Export儲存為FASTA、Text、HTML及其他的檔案格式,下圖是儲存為HTML的檔案在瀏覽器開啟的結果。
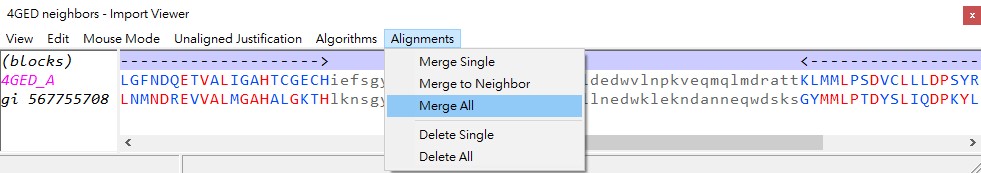
前面介紹以Cn3D來查看VAST預先計算完成的疊加結構及其對應的序列排列,Cn3D也可以輸入序列及三維結構進行比對。回到原先下載的疊加結構及其對應的序列排列檔案(包括4GED_A、5EJT_A、3FJW_A及1IYN_A),選擇序列排列視窗中的Imports→Show Imports開啟Import Viewer,現在加入(Merge)一個從網路或已下載結構的步驟如下:
1. 在Import Viewer中選擇Edit→Import Structure→From a File→開啟mmdb_4NVB.cn3,序列加入後均呈現未排列排列的灰色小寫字型。
2. 選擇Algorithms→Block Align N→出現Set Block Aligner Options設定→點擊OK,藍色大寫字型的排列區域即出現。
3. 選擇Alignments→Merge All,4NVB(gi567755708)的序列加入序列視窗,並與其他四個序列排列如下圖。
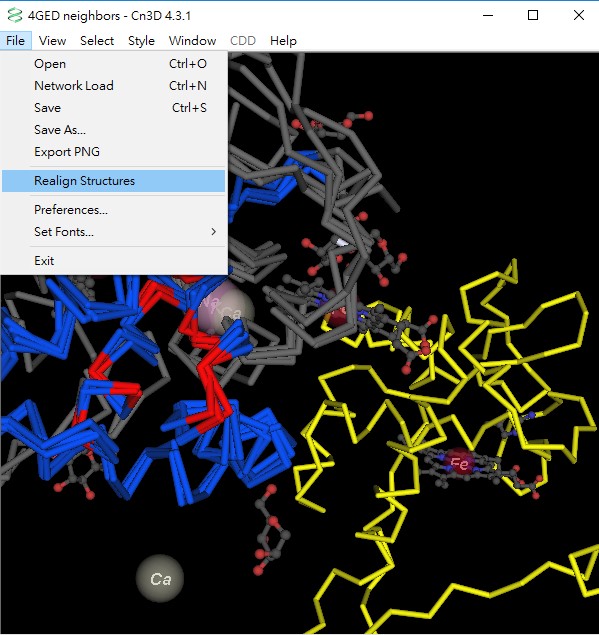
4. 此時在結構視窗中還無法看見4NVB的結構,所以必須先選擇File→Save As,儲存為另一個cn3的檔案。
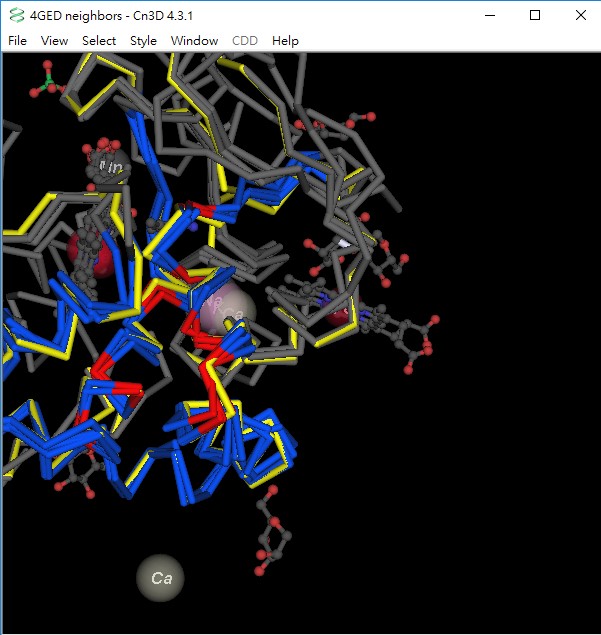
5. 然後開啟此一新檔案,在結構視窗中黃色突出的部分即為4NVB的結構,但是它還未與其他結構疊加一起。選擇File→Realign Structures後,它則移位至其他結構並疊加一起。
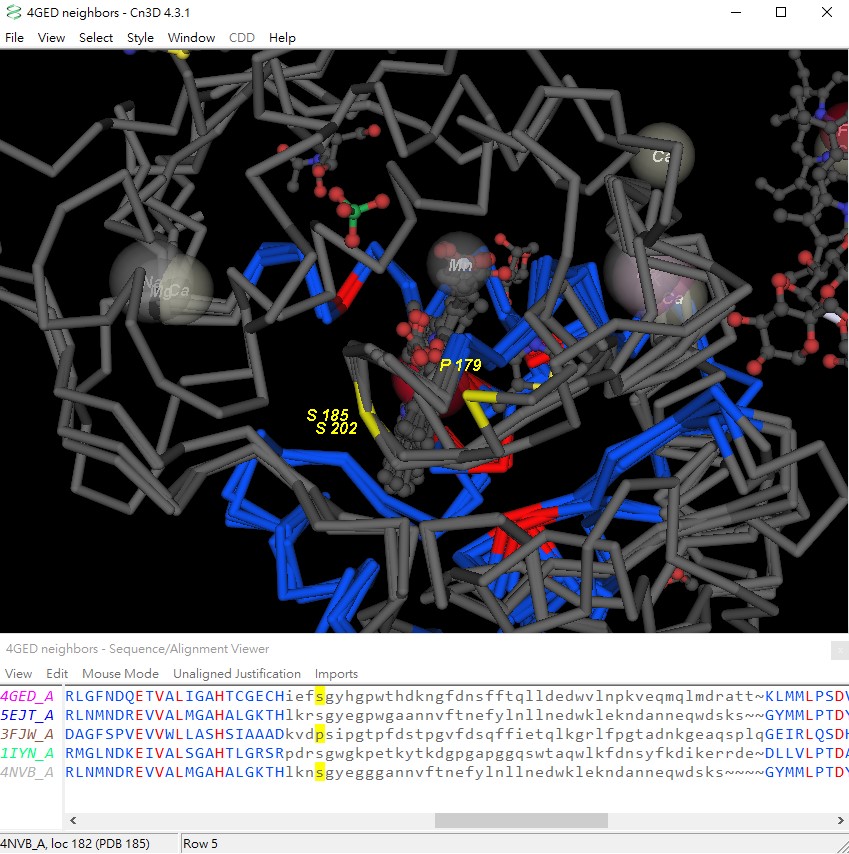
突出Ser202在4GED_A的位置在序列視窗選擇Mouse Mode →Select Rectangle,點擊4GED_A序列中的S202後,按壓Shift+點擊同一列上的p(3FJW_A)和s(4NVB_A)。在結構視窗選擇Style→ Annotate,在User Annotations中點擊New,並填入Ser202→Edit Style,在Style Options表單的Labels中Protein backbone項目的Spacing為1、Type為One Letter及Numbering為From PDB。如此可知Ser202在4GED_A相對於4NVB_A是Ser185。
Cn3D是具有許多功能的軟體,除了上述的操作入門,使用者若有其他感興趣的部分,請參考Cn3D (https://www.ncbi.nlm.nih.gov/Structure/CN3D/cn3dtutP1.shtml) 網頁中的教學課程或Cn3D的幫助文檔,選擇Help→Commands即可。
感謝生醫所黃明經老師實驗室孫慶姝小姐提供以上寶貴資料