本文經訊聯基因數位股份有限公司同意轉載
關於IPA與IPA Interpret
QIAGEN IPA是一款雲端生物資訊分析軟體,可用來分析以下多種實驗數據:
- RNA-seq表現量資料
- 基因晶片(microarray)
- SNP陣列
- 蛋白質體學
- 代謝體學
- 其他產生基因清單的小型實驗(如PCR)
除了資料分析,IPA還能搜尋特定基因、蛋白質、化學物質、疾病與藥物的資訊,建立專屬的生物模型。透過IPA的分析和建模功能,再搭配結構化整理的生物與化學研究發現資料庫,能幫助您更加了解研究數據在更大的生物或化學系統中的意義。
IPA Interpret為IPA中包含的元件,可透過快速且現代化的網頁介面呈現IPA Core Analysis的結果,並與他人分享。
以泡泡圖(bubble chart)精進您的數據解釋與呈現
IPA Interpret新增功能-泡泡圖,能協助您:
- 快速地找出分析結果中有趣的模式
- 聚焦於最重要的預測
- 產生適用於簡報或發表文獻的視覺化圖表
泡泡圖適用於Canonical Pathways、Upstream Regulators、Diseases & Functions,以及Tox Functions等分析,並可在主頁相關項目的「View details」連結進入的Bubble Chart分頁中查看。
泡泡圖呈現
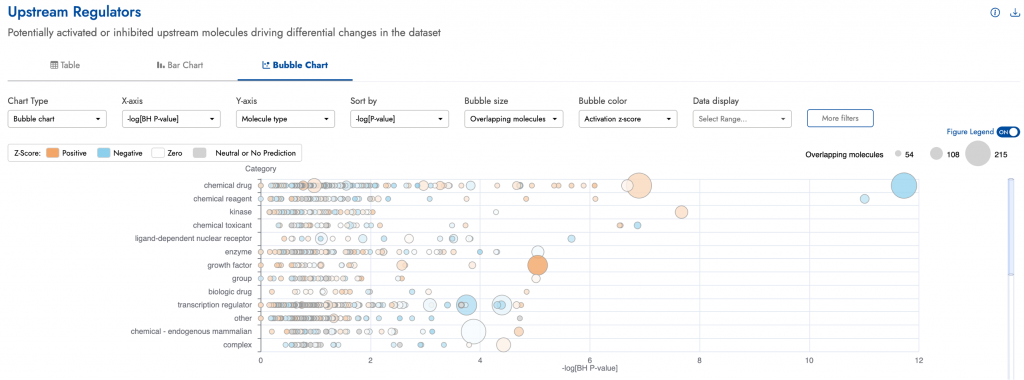
圖一顯示了IPA Interpret畫面中以分類泡泡圖呈現的Upstream Regulator分析結果。圖中Y軸為調控因子的「分子類型」,X軸則是每個調控因子以Benjamini-Hochberg多重檢定校正後p值的負對數,來顯示它的顯著程度。這種視覺化方式能讓您快速看出在此資料集中,預測最顯著的調控因子(圖表右側)主要為化學藥物或化學試劑。
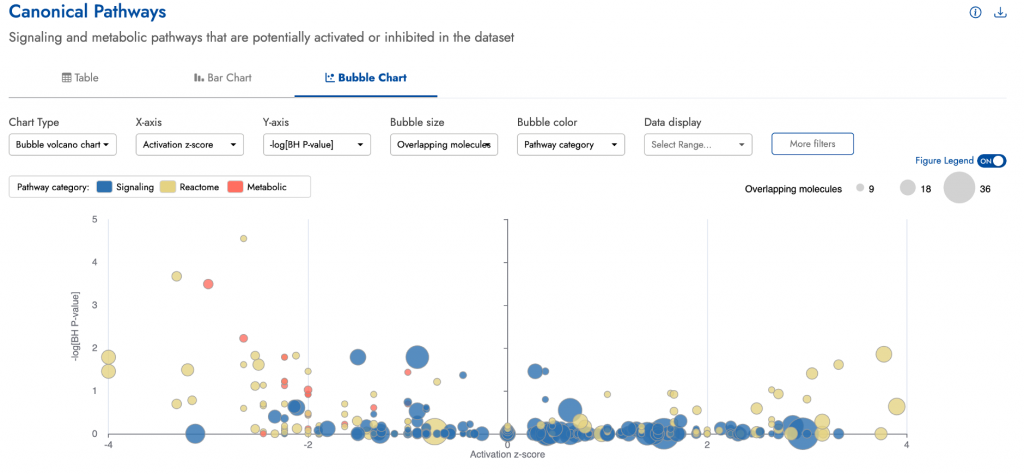
以火山圖風格展示泡泡圖
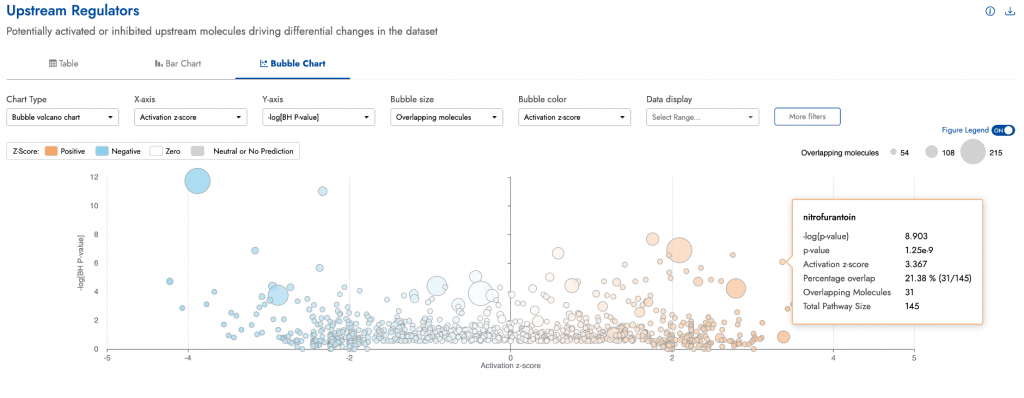
您可以依分數、調控因子名稱甚至是根據整個類別篩選結果(見圖五)。也可以將結果改用「火山圖風格」的泡泡圖顯示。圖二顯示了與圖一相同的結果,但以火山圖呈現,Y軸為顯著性(BH p值),X軸為活化方向(activation z-score)。
細節頁面新增「Expected」欄位
將游標移到泡泡上時,會顯示該泡泡的評分指標摘要(見圖二)。點擊泡泡後,圖表下方會顯示更多細節,包括路徑或網路圖、該項目的文字說明,以及資料集中支持該預測的基因詳細列表。
在詳細列表中,新增了一欄「Expected」,其數值表示若該項目(如上游調控因子、Canonical Pathway等)被活化時,資料中的基因或蛋白應呈現的調控方向(上調或下調)。這些資訊是根據文獻資料整理而來。
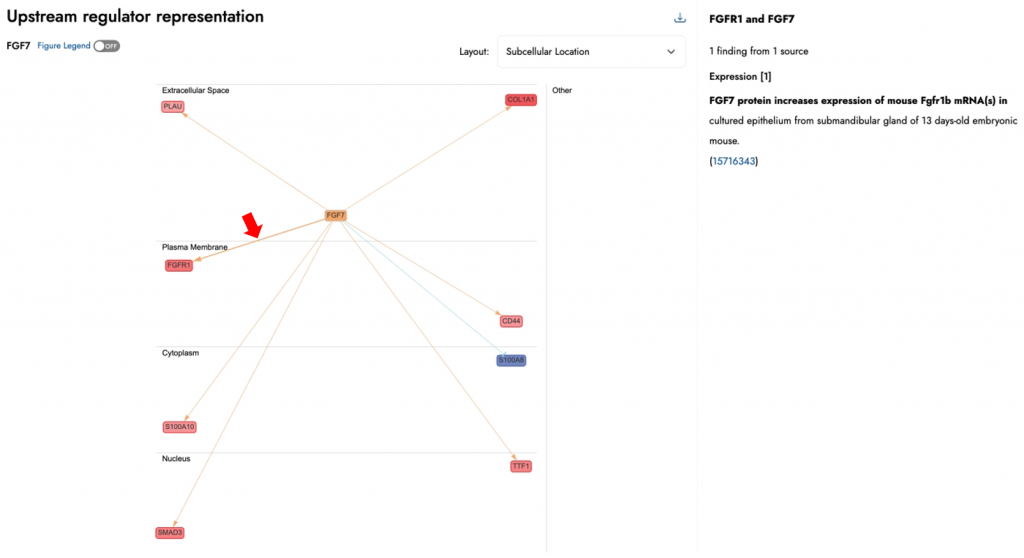
圖三示範了在特定資料集裡,上游調控因子FGF7的分析結果。其中有七個受FGF7調控的基因預期會上調,一個預期下調,如Expected欄所示(紅框)。資料集的相鄰欄位「Expr Log Ratio」(藍框)顯示這八個基因的表現方向都與預期一致,因此在此分析中FGF7被預測為活化(z-score = 2.83)。

此外,如圖四所示,您可以點選網路中的關係連線,來查看支持Expected欄位資訊的相關研究結果。
使用More filters進行額外篩選
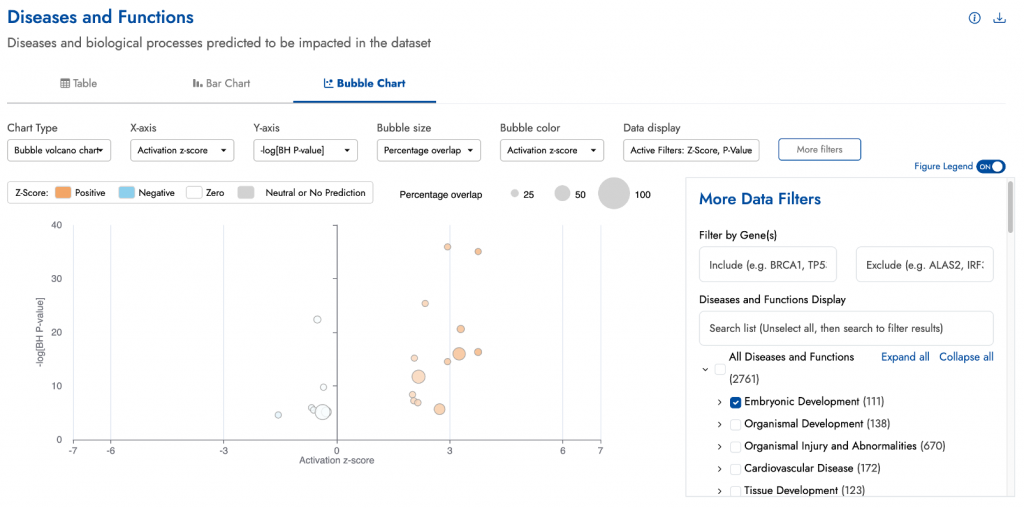
不同類型的「項目」都有專屬的分類方式,例如Upstream Regulators和Tox Functions的分類就不一樣。圖五示範了用「More filters」選項,將疾病與功能篩選成胚胎發育(Embryonic Development)類別,方便您挑選適合發表或簡報的重點結果。
按照路徑分類篩選與上色泡泡
此外,Canonical Pathway的泡泡圖可以依「路徑類別(Pathway Category)」上色(見圖六),顏色分別對應到訊息傳遞、Reactome資料庫或代謝路徑。您也可以選擇依路徑類別進行篩選,隱藏其中一個或多個類別的所有泡泡。
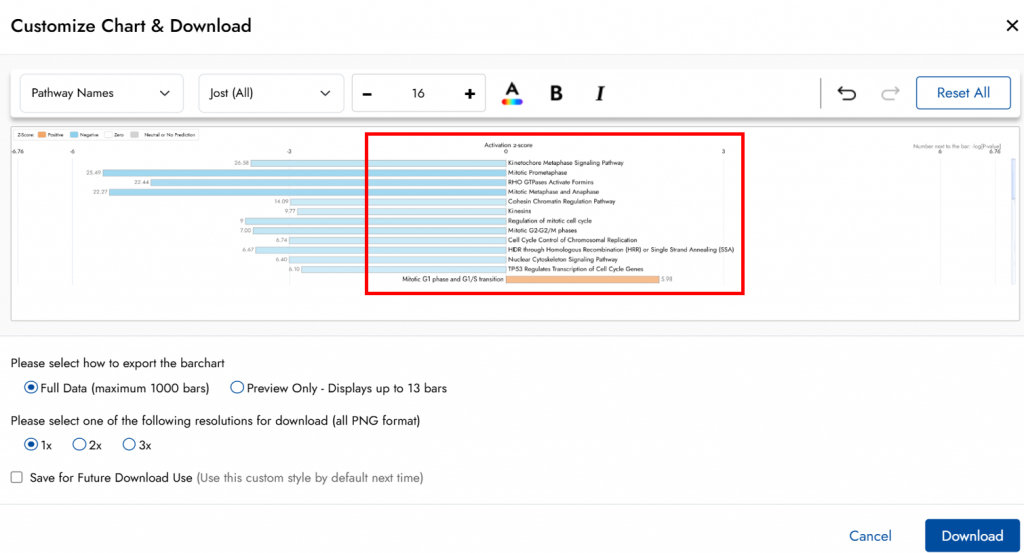
匯出自訂的長條圖
IPA Interpret現在支持在匯出圖片時,對長條圖進行更完整的自訂設定。您可以輕鬆調整多個選項,包括:
- 字體
- 長條間距
- 長條粗細
- 長條配色
您可以依照以下步驟操作這項功能:
- 篩選想要顯示的長條
- 點選匯出(export)按鈕
- 在出現的選單中選擇「Custom styling before download」
- 在彈出的視窗中,用各種控制項調整圖表,打造符合需求的輸出格式
若想保存這個新格式,勾選視窗底部的「Save For Future Download Use」,之後下載相同類型的圖表就能沿用設定。如果想恢復預設樣式,隨時可點選「Reset All」按鈕。

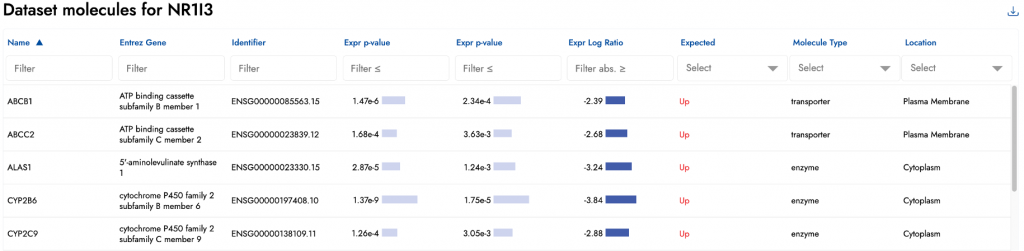
圖形摘要功能強化
IPA Interpret的「Graphical Summary」現在經改版後更容易解讀,您可以直接查看摘要圖中預測的上游調控因子對應的基因表現細節。從主頁點選「View details」後,將游標移到Graphical Summary 網路裡感興趣的上游調控因子上,就會跳出提示文字(tooltip),顯示該基因在分析中的分數,以及其在資料集中的表達詳細資訊,方便您同時評估基因表現與調控預測。
例如圖四中,游標停在NR1I3(又稱CAR)節點上時,可看到它預測為受抑制,且在資料集中為下調表現。不過這個基因本身未通過本次分析設定的p值<0.01的篩選(提示文字中顯示「Passed Cutoffs No」)。
此外,點選Graphical Summary中的任意項目後,下方會顯示一張表格,列出資料集中用於預測該項目活性的所有基因。圖八下方就顯示了與NR1I3相關聯的基因列表。
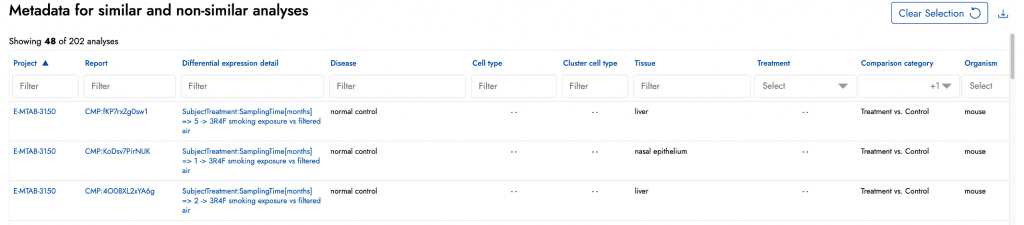
透過新增的詮釋資料表(Metadata Table)精進Similar Analysis圖表的探索功能
現在,您可以對 IPA Interpret中「Similar Analyses」圖表裡約200筆分析的詮釋資料(metadata)詳細資訊進行排序和篩選,讓您更容易縮小範圍找到感興趣的分析。舉例來說,您可以只看「Treatment vs. Control」類型的分析。圖九展示了這種方便的篩選功能。
新增API支援程式化提交分析並取得IPA Interpret連結
IPA原本就有提供API讓使用者可以在不開啟桌面版的情況下,透過程式自動將資料集提交至雲端進行分析,並附有R和Python範例腳本方便您試用APIs或將其整合到自動化流程中。
本次更新新增了一個API,可讓您的程式在分析完成後自動取得並開啟IPA Interpret的報告連結。也就是說,您可以先程式化提交資料,幾分鐘後就能在瀏覽器查看分析結果,或將網址儲存以後使用。詳細使用方式可參考API說明頁、範例腳本和PDF手冊。
IPA Interpret其他改進內容
- 長條圖中的工具提示會在游標移到長條上時顯示該項目的詳細資訊
- 資料表新增了列計數器,可顯示表格的總列數,以及篩選後剩餘的列數
- 介面中現在會以「AI-Suggested」標誌標示由大型語言模型產生的摘要內容
- 新增了彈出式說明面板,用來解釋項目表中的z-score
IPA桌面版:路徑與網路即時因果評分(Grow 工具)
現在IPA 桌面版中的Build > Grow工具已更新,可更快速分析任意路徑或網路,找出可能造成因果影響(或受其影響)的相關上游調控因子、Canonical Pathways、疾病/生物功能或毒理功能,並即時進行預測與評分,加快探索與建立假說的流程。例如,您可以做以下探索:
- 找出路徑的上游化學抑制劑
- 推測某個路徑活化後可能被抑制的其他路徑
- 分析上游調控如何驅動下游結果
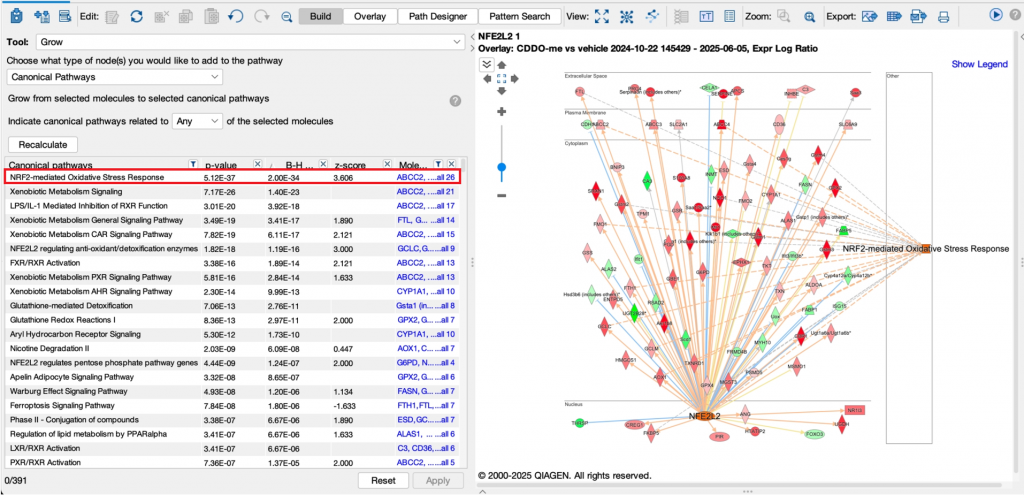
實際應用(一)
在查看分析結果中的上游調控因子時,您可能想知道哪些Canonical Pathways會受影響,圖十示範了這樣的分析流程:
- NFE2L2在分析中預測為活化,畫布上以細胞內模式(Subcellular mode)顯示它和它的調控基因的關係
- 使用Build > Grow > Canonical Pathways功能,預測此網路中可能活化或抑制的路徑
- 結果顯示NRF2調控的氧化壓力反應路徑預測為活化,z-score為3.606(見紅框)
- 選取該路徑後,會自動加到畫布中並以橘色標示其為活化狀態(圖右側)
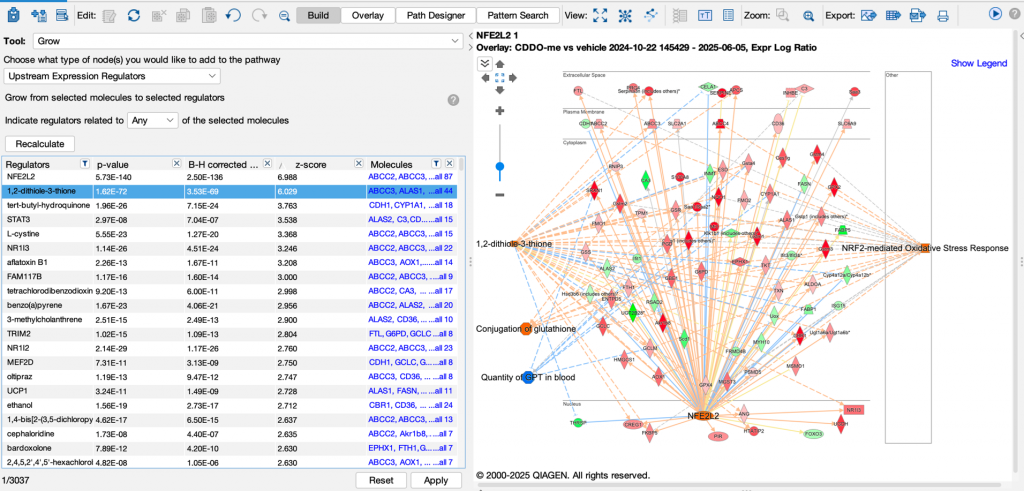
您還可以進一步加入其他類型的項目,如圖十一所示,左側網路中新增了Grow工具建議的Tox Function、Bio Function和上游調控因子。
活動預測需要路徑上的分子有顏色標示(紅色或綠色),用來表示表現量或磷酸化方向。這些顏色可以來自套用的分析結果、資料集,或是您以MAP工具手動上色。如果沒有這些顏色標示,Grow工具就只能提供p值預測,而無法計算活化狀態。
實際應用(二)
您還可以用更新後的Grow工具來探索其他問題,例如找出調控特定Canonical Pathway的因子。圖十二示範了這個方法:
- 開啟「T細胞訊號傳導中的全身性紅斑狼瘡(SLE)」路徑
- 套用MAP工具或按鍵快捷方式(a鍵)顯示其節點的活化模式
- 選擇Build > Grow > Upstream Expression Regulators
- 依z-score排序表格
可以看到匹格列酮(pioglitazone)為「受抑制」的調控因子(負的z-score),表示相反來說它可能對SLE產生抑制作用。
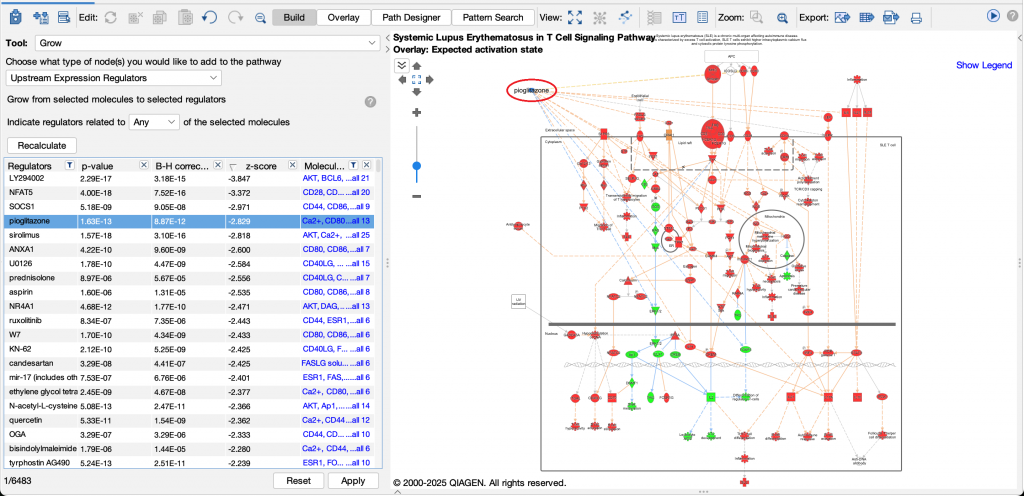
套用Canonical Pathway節點的活化模式。匹格列酮(紅框)在此路徑呈現負的z-score,顯示它可能對全身性紅斑狼瘡(SLE)具有治療潛力。
此外,您可以先在路徑或網路上選取特定節點子集,再只對這些節點套用 Grow工具。這種做法能幫助您找到更具局部相關性的連結。
內容更新
新增六條 Ingenuity 信號轉導通路:
- DYRK1A訊號傳導路徑(DYRK1A Signaling Pathway)
- GAIT轉譯調控訊號傳導路徑(GAIT Translation Signaling Pathway)
- 減數分裂起始訊號傳導路徑(Meiosis Initiation Signaling Pathway)
- 膜修復訊號傳導路徑(Membrane Repair Signaling Pathway)
- NF1介導的RAS訊號傳導路徑(NF1 RAS Signaling Pathway)
- 應激顆粒訊號傳導路徑(Stress Granule Signaling Pathway)
知識庫更新
新增的38萬多筆新發現,使知識庫總數達到1,460萬筆。
- 超過249,000筆來自專家文獻註解
- 超過10,000筆BioGrid蛋白質交互作用資料
- 超過16,000筆基因與疾病的GWAS研究發現
- 超過109,000筆ClinVar癌症突變資料
- 超過1,200筆ClinicalTrials.gov藥物與疾病資料
- 超過1,300筆 ClinicalTrials.gov靶點與疾病資料
- 超過2,100筆基因本體(Gene-Ontology)研究發現
新增兩個物種的資料集對照
支援上傳以下物種的資料集:
- 山羊(Capra hircus)
- 三刺魚(Gasterosteus aculeatus)
OmicSoft資料集
OmicSoft中新增了15,001個資料集,使總資料集數達到246,776。另外,此版本中新增了一個新資料來源名為:美國國家癌症研究所患者來源模型(NCI Patient-Derived ...)。這些資料集將於七月提供。
| Land | Repository | Q1 | Q2 | Increase |
| DiseaseLand | HumanDisease | 38,710 | 39,036 | 326 |
| MouseDisease | 28,784 | 29,355 | 571 | |
| RatDisease | 10,264 | 10,269 | 5 | |
| LINCS | 25,880 | 25,880 | ||
| -OncoLand | -OncoHuman | 24,972 | 25,262 | 290 |
| -OncoMouse | 1,516 | 1,516 | ||
| TCGA | 4,854 | 4,854 | ||
| ENCODE RNA Binding | 486 | 486 | ||
| ClinicalProteomic Tumor | 2,978 | 2,978 | ||
| NCI Patient-Derived Models | 552 | 552 | ||
| Single Cell Land | SingleCellHuman | 194 | 194 | |
| SingleCellHumanUmi | 77,140 | 81,140 | 4,000 | |
| SingleCellHumanHCL | 1,469 | 1,469 | ||
| SingleCellMouse | 81 | 81 | ||
| SingleCellMouseUmi | 13,135 | 22,392 | 9,257 | |
| Normal Cells and Tissues | Human Tissues (GTEx) | 1,312 | 1,312 |
原始文章來源:Latest improvements for IPA