文:張耀明研究助技師/生物醫學科學研究所
如果經過生物實驗後我們有幾組「有顯著表現差異基因(differentially expressed genes, DEGs)」的列表,我們想要知道這些基因可能參與在哪些類型的功能(gene ontology, GO)或是生物途徑(biological pathway)裡,有什麼免費方便的工具可以幫我找出來呢?
今天我們介紹的是一個好用的線上資料庫網站DAVID (The Database for Annotation, Visualization and Integrated Discovery),雖然它是一個免費的網路服務,但它的功能已經相當完整一點也不輸給許多商用軟體。
首先,我們要先準備一組基因列表(a list of Gene IDs),列表中通常是在實驗結果中有顯著差異的基因(DEGs),而DAVID網站支援很多不同資料庫的Gene ID格式,如常用的ENSEMBL gene ID或是Affymetrix microarray 的probe ID等。此外,為了透過統計的方式找出DEGs參與的生物功能(biological functions)或是路徑(pathways),我們通常也會準備另一組基因列表當作背景資料(background data),這組基因列表通常是實驗中「所有表現基因(all expressed genes)」的列表。如果沒有,DAVID網站也會根據不同的物種提供「所有註解基因(all annotated genes)」列表來當作預設的背景資料。
資料準備好之後我們接下來就一步步告訴大家如何使用:
一、在網頁瀏覽器上開啟連結進入DAVID網站(https://david.ncifcrf.gov/home.jsp),接著在網頁上方的橫幅功能選單上點選 “Start Analysis”
圖一、DAVID網站首頁
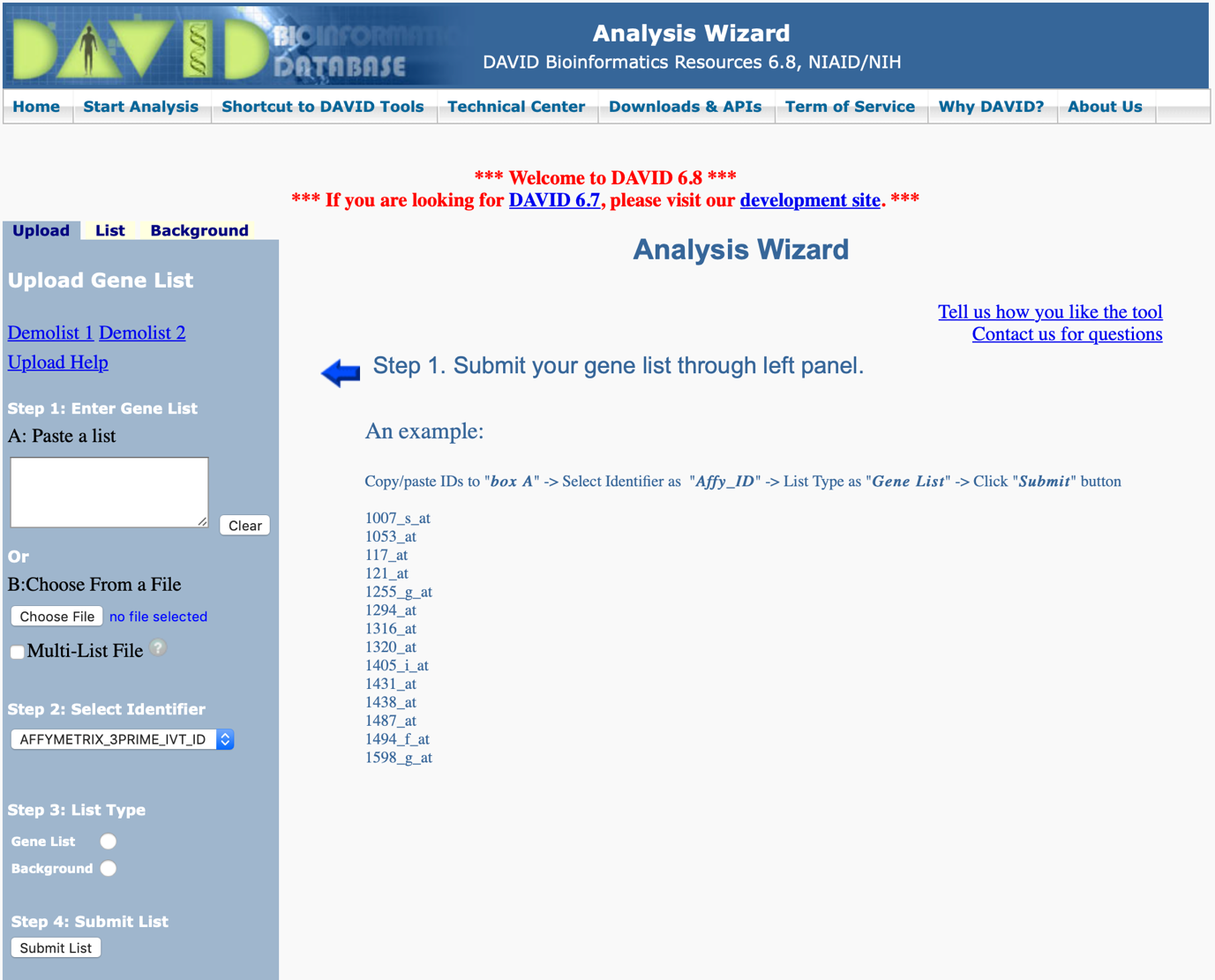
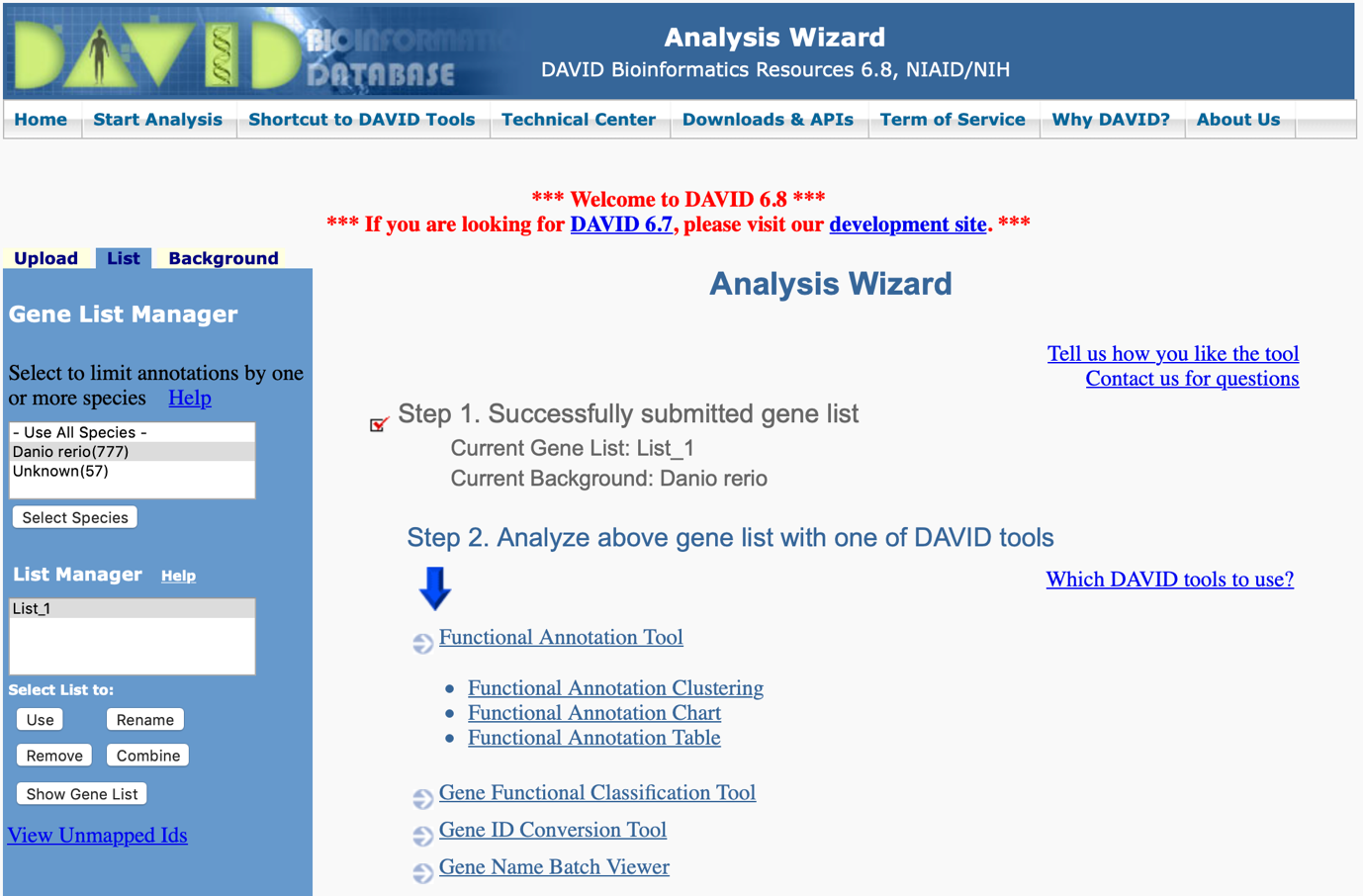
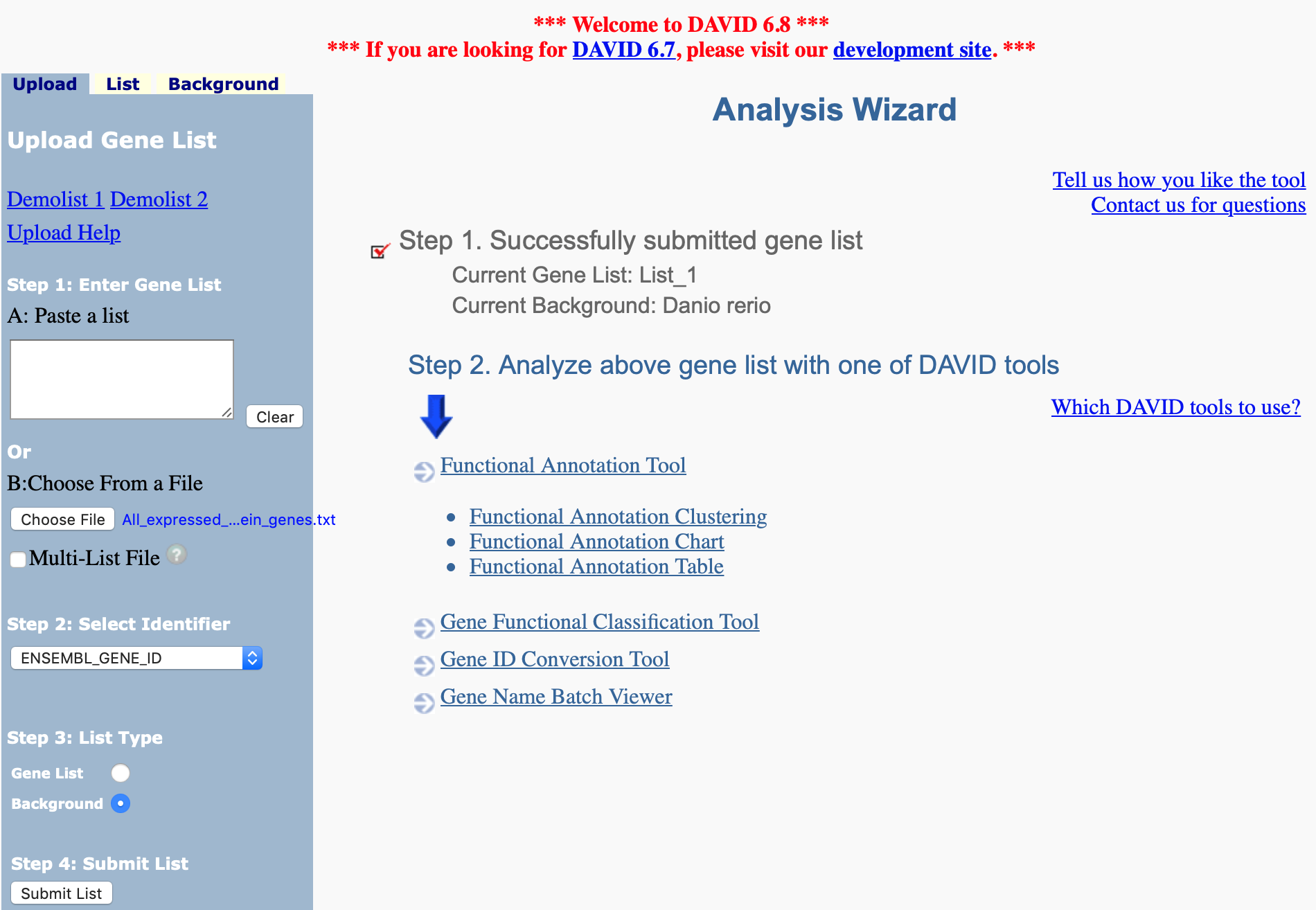
二、在網頁上的左邊會出現可以讓我們上傳基因列表的表單,我們可以直接利用複製貼上的功能直接將基因列表貼在A: Paste a list下方的空白處,接著在表單Step 3: List Type的地方要點選Gene List後並按下 “Submit List” 按鈕(圖二、三)。另一個方式則是先將基因列表儲存成文字檔案,利用B: Choose From a File的按鈕選擇要上傳的基因列表(下一個步驟會示範)。上傳成功後,原來的上傳表單會出現List Manager並顯示上傳的基因列表編號(圖四中的List_1)。
圖二、點選Start Analysis後,畫面左邊出現可上傳基因列表的表單
圖三、將基因列表貼在A: Paste a list下方的空白處
圖四、上傳完成後,List Manager會出現一筆資料(List_1)
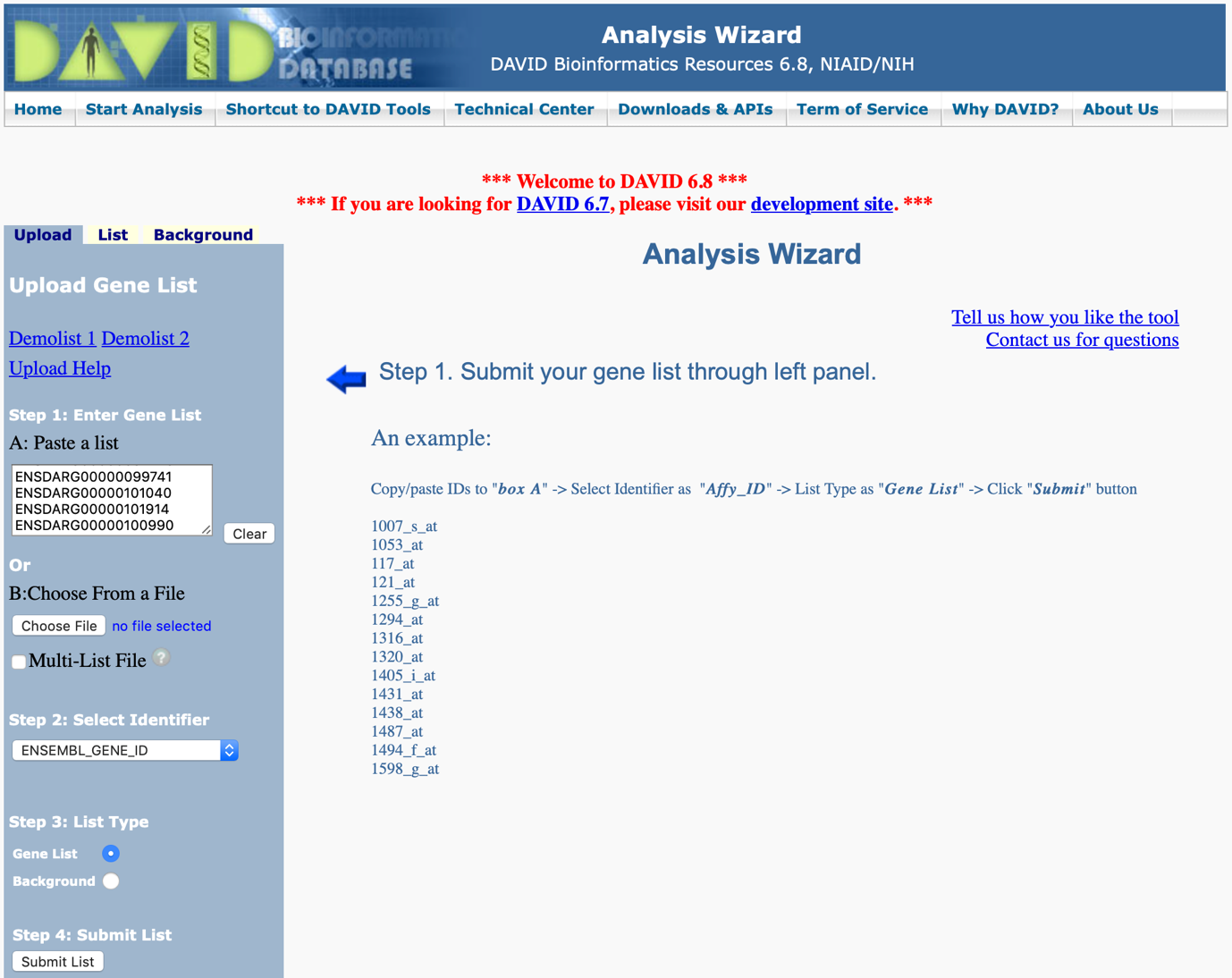
三、接著我們再上傳一組背景資料用的基因列表。請點選左邊表單中 “Upload”,這次利用另一個上傳方式B. Choose From a List來上傳基因列表。按下 “Choose File”按鈕,然後選取我們要上傳的檔案後,按鈕旁邊會出現選擇檔案的名稱。此時因為我們的基因列表是要拿來當作背景資料,所以記得要在Step 3: List Type的地方選擇Background後按下 “Submit List” 按鈕(圖五)。值得注意的是,因為網路頻寬的大小因素,有時候會因為上傳的基因數量太多,上傳的速度會有點慢,請稍微耐心等候。
圖五、利用檔案來上傳基因列表
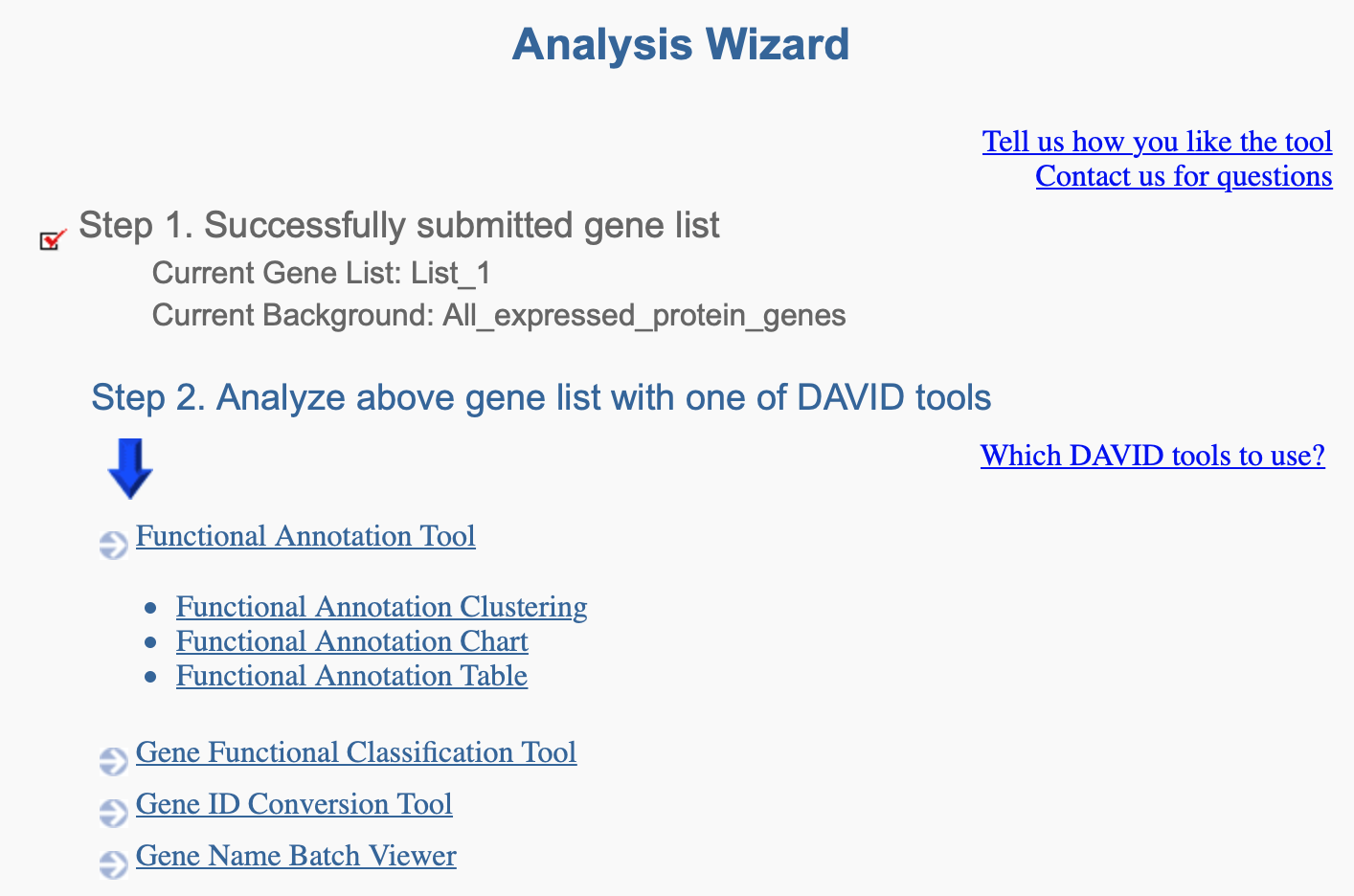
圖六、資料上傳完成後,出現開始分析的畫面
四、當資料都上傳完成後,網頁的中間部分會出現可以開始分析的畫面(圖六),這時依照箭頭指示點選 “Functional Annotation Tool” 的連結即可。
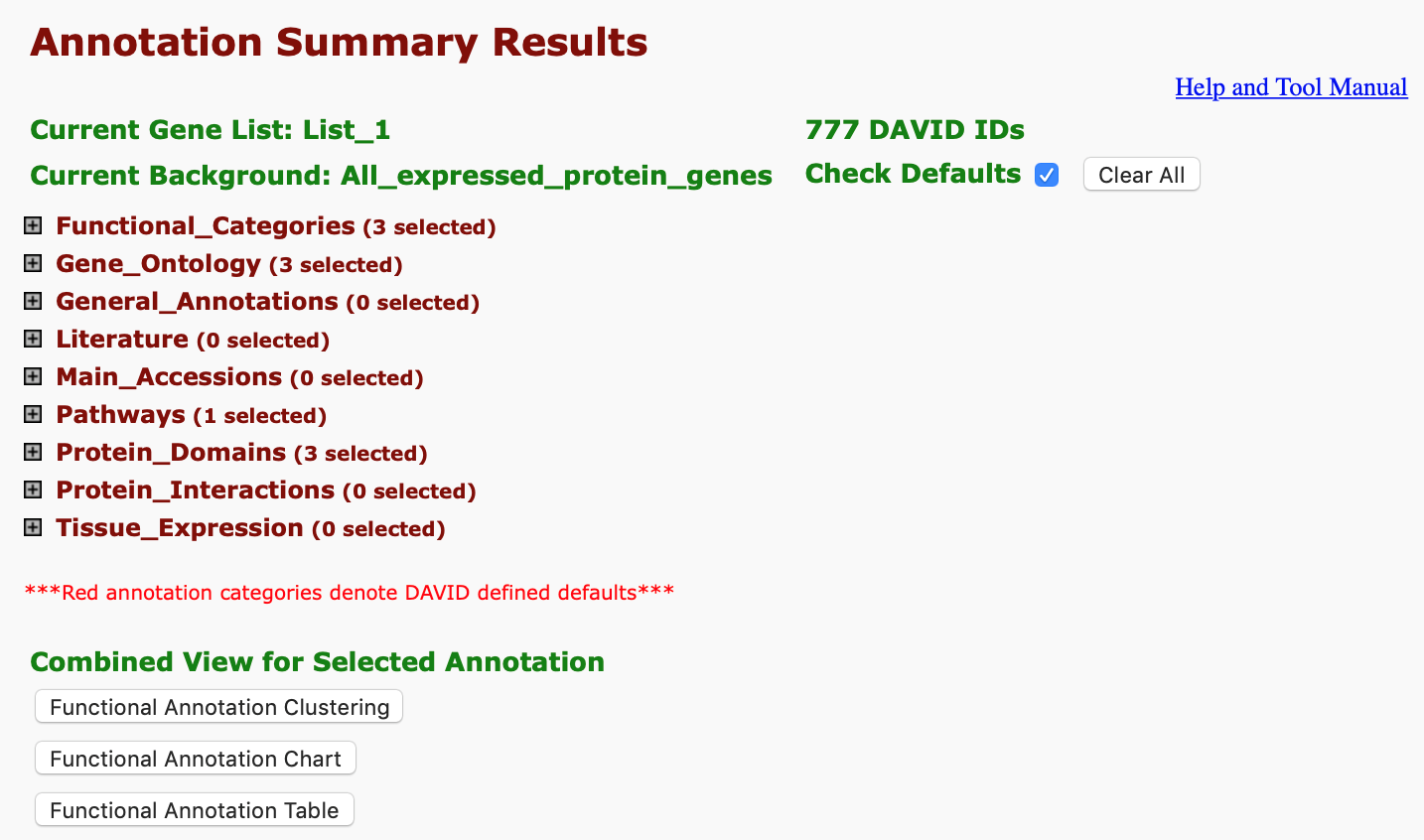
圖七、分析完成,讓使用者點選結果類別的畫面
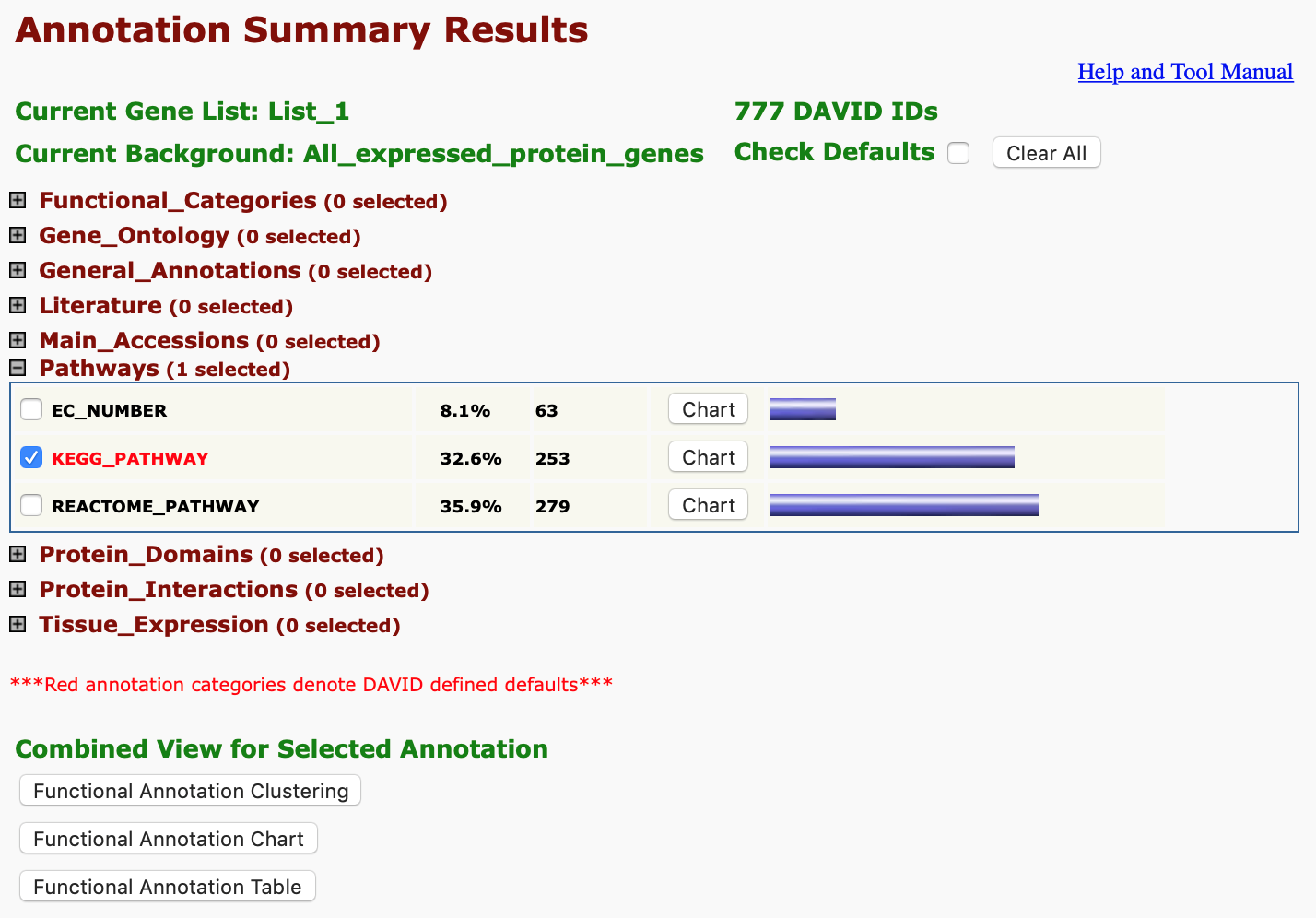
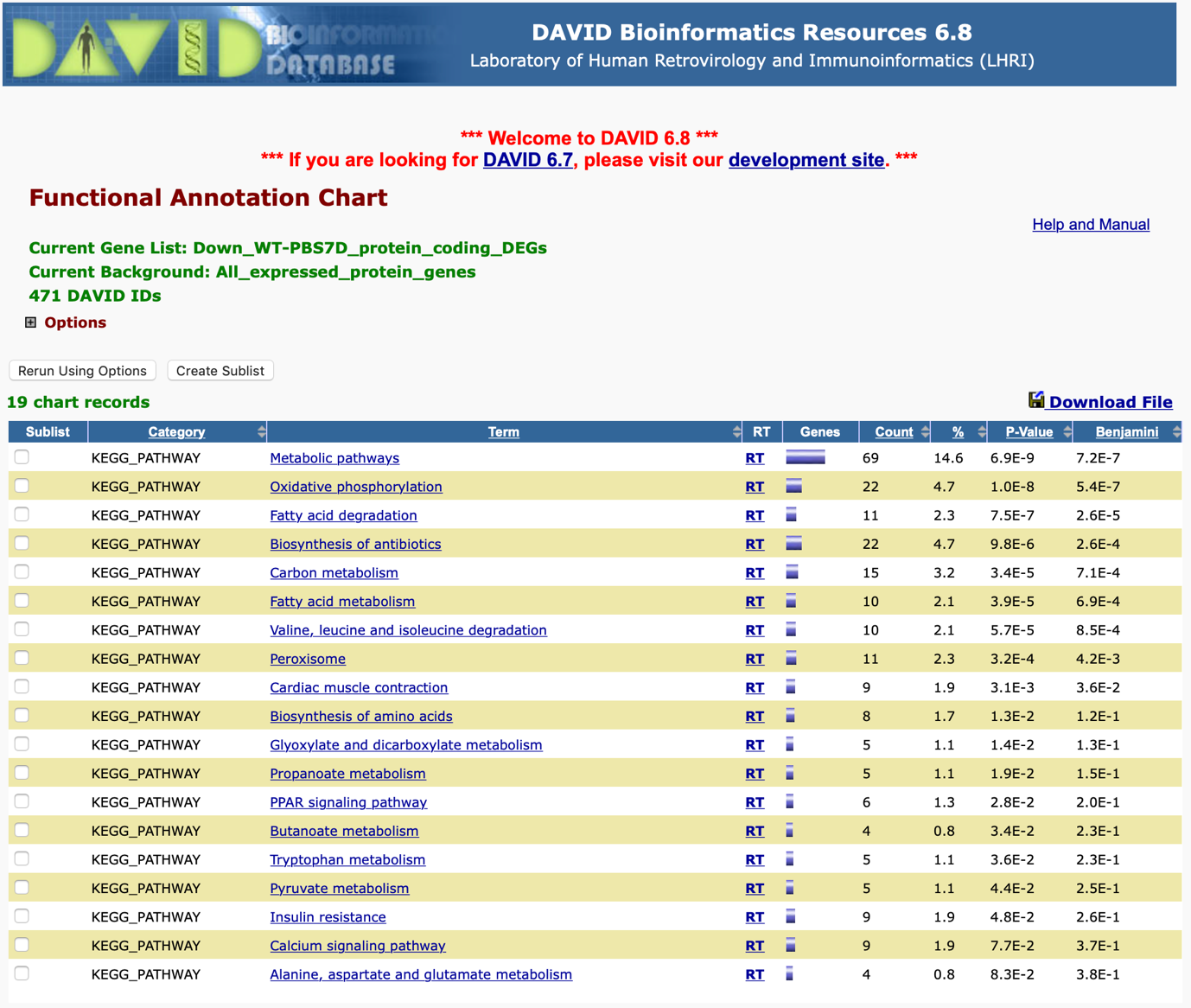
五、當分析完成,DAVID可以讓我們選擇我們想要看哪些結果(圖七)。由於預設的功能或是路徑類別有相當多,不一定都符合我們想要看的類別,所以可以先點選右上角 “Clear All” 的按鈕,然後再依照自己的需求點選類別。例如我們關心的是 DEGs列表的基因在哪些KEGG pathways中扮演重要的角色?這時我們就點選Pathways中的KEGG_PATHWAY 後再按下方 “Functional Annotation Chart” 按鈕。
圖八、清除預設選擇,單獨選擇KEGG_PATHWAY
六、分析結果會以表格的方式顯示(圖九),除了參與生物路徑的名稱之外,各項統計的資料也會列出來。如果我們要更完整的結果可以點選右上角 “Download File”的連結。
圖九、結果顯示我們上傳的基因中參與了許多不同的pathways,每一個pathway的統計資料也一併列在右邊的欄位中。
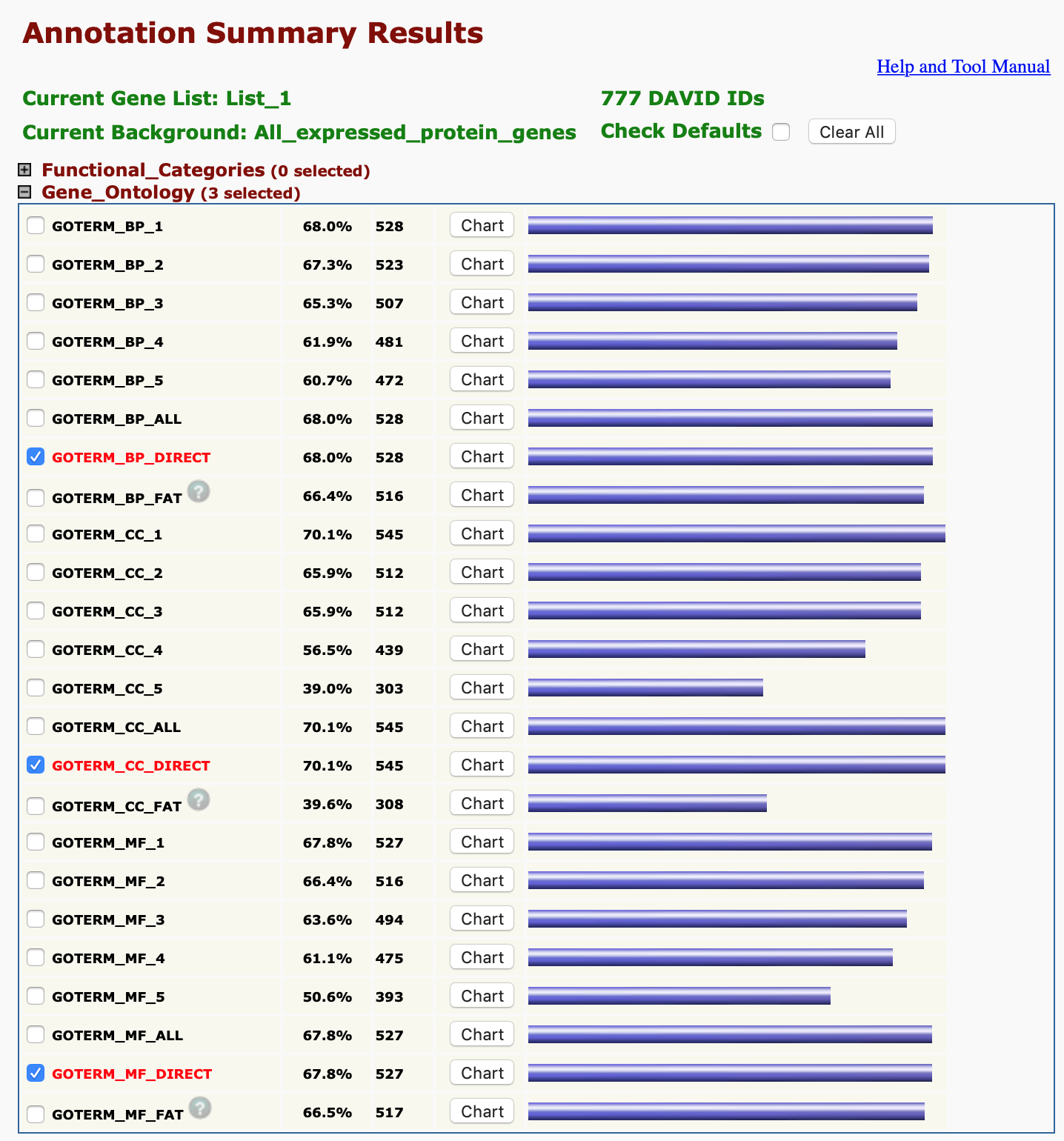
七、同樣的,如果我們想要看這些基因參與的GO function,我們也可以選擇類別的畫面選擇Gene_Ontology中GOTERM_BP_DIRECT、 GOTERM_CC_DIRECT以及GOTERM_MF_DIRECT類別(圖十)之後再按下方 “Functional Annotation Chart” 按鈕即可。
圖十、清除預設選擇,單獨選擇Gene_Ontology的類別