文:張耀明研究助技師/生物醫學科學研究所
在基因表現分析流程中,我們找出實驗組與對照組有差異的基因(DEGs; differentially expressed genes)之後通常會接著進行FEA (Functional Enrichment Analysis)去看看他們是否特別出現在哪些生物功能或是途徑上(overrepresented functions or pathways)。不過我們常常遇到的問題是,得到的功能類別或途徑似乎與我們實驗觀察到的表現型(phenotype)沒什麼關聯。換句話說就是我們預期想看到的功能都沒有出現在分析結果裡面,怎麼辦呢?其實這個時候我們可以反向思考,先找出我們有興趣的功能類別或途徑所組成的基因群(gene sets),然後檢查這些基因在實驗組與對照組是否有差異表現的趨勢?而這樣的分析就稱為GSEA (Gene Set Enrichment Analysis)。
先前我們跟大家介紹過好用的DAVID網站,只要上傳DEGs清單,我們就可以找出相對應的生物功能(biological functions)或是路徑(pathways)。但是這個分析方法必須仰賴DEGs,而決定DEGs方法通常有很多不同的方法與標準,進而造成結果的差異。這一次我們要介紹的GSEA則提供了另一種分析方法,它避免了先決定DEGs的問題,且跟FEA方式相反,GSEA是從已經定義好參與某些功能的基因群(gene sets)出發,然後檢查這些基因的表現量是否在不同的樣本間有特別的差異趨勢。
事實上,網路上有不少工具都提供了GSEA分析,但其中Broad Institute提供的軟體功能最為完整,以下就是基本的分析流程步驟:
一、下載Broad Institute GSEA工具程式
二、執行GSEA程式
三、準備輸入資料
四、匯入資料
五、選擇Gene sets
六、設定比對群組
七、選擇對應表
八、進行分析
一、下載GSEA工具程式
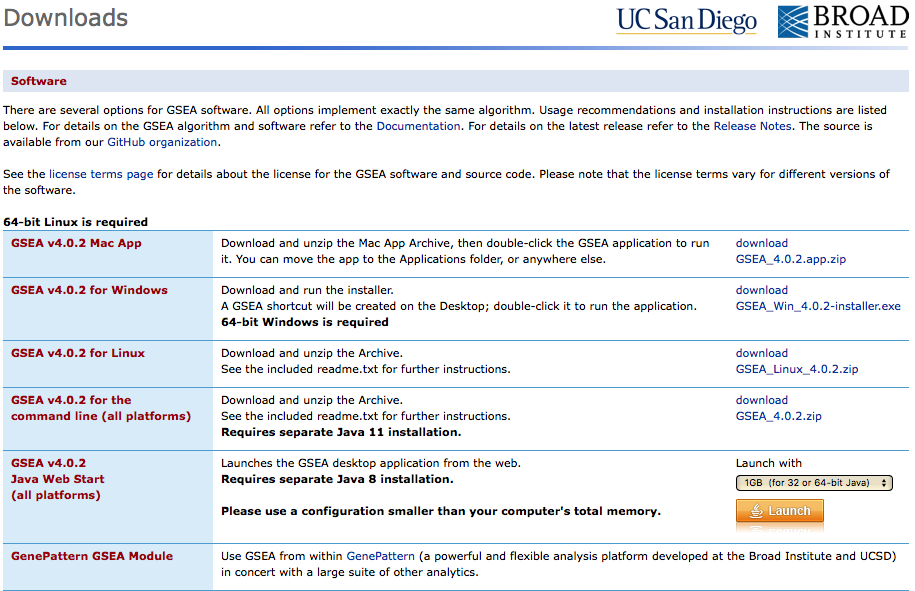
首先我們到http://software.broadinstitute.org/gsea/index.jsp網站去下載軟體(圖一)。
圖一、Broad Institute 的 GSEA網頁
點選download之後,會出現一個註冊的頁面,由於這個軟體提供學術研究免費使用,所以填寫email和基本資料就可以進入下載頁面了(如圖二)。
圖二、請根據自己的電腦作業系統選擇對應的版本。
二、執行GSEA
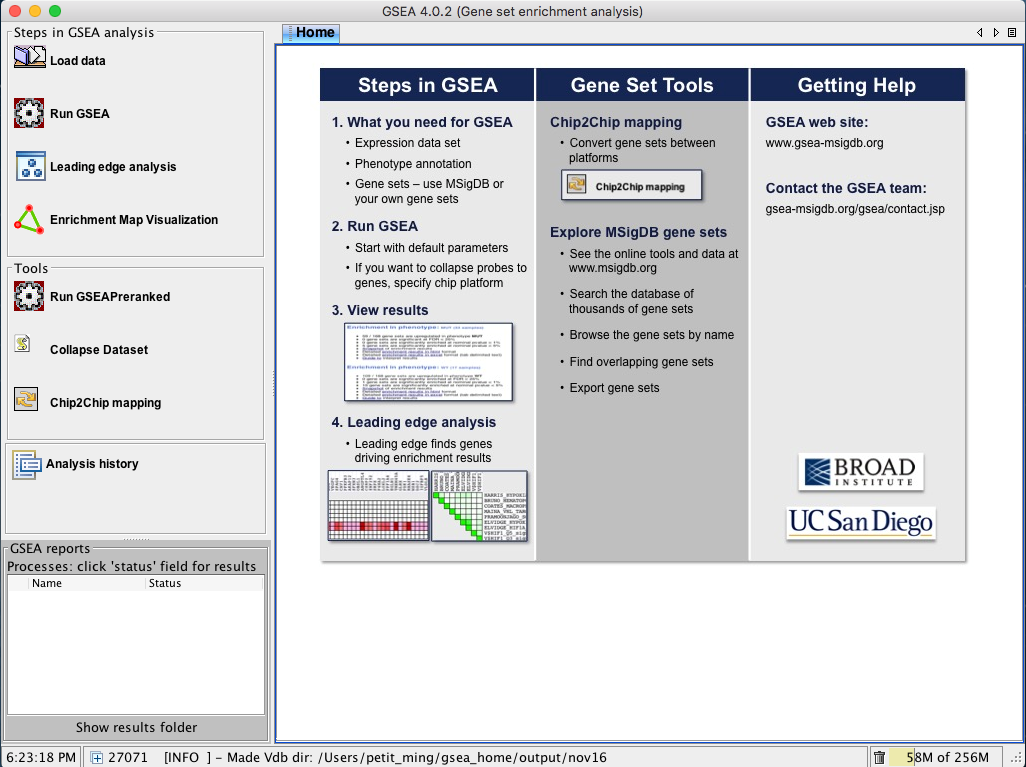
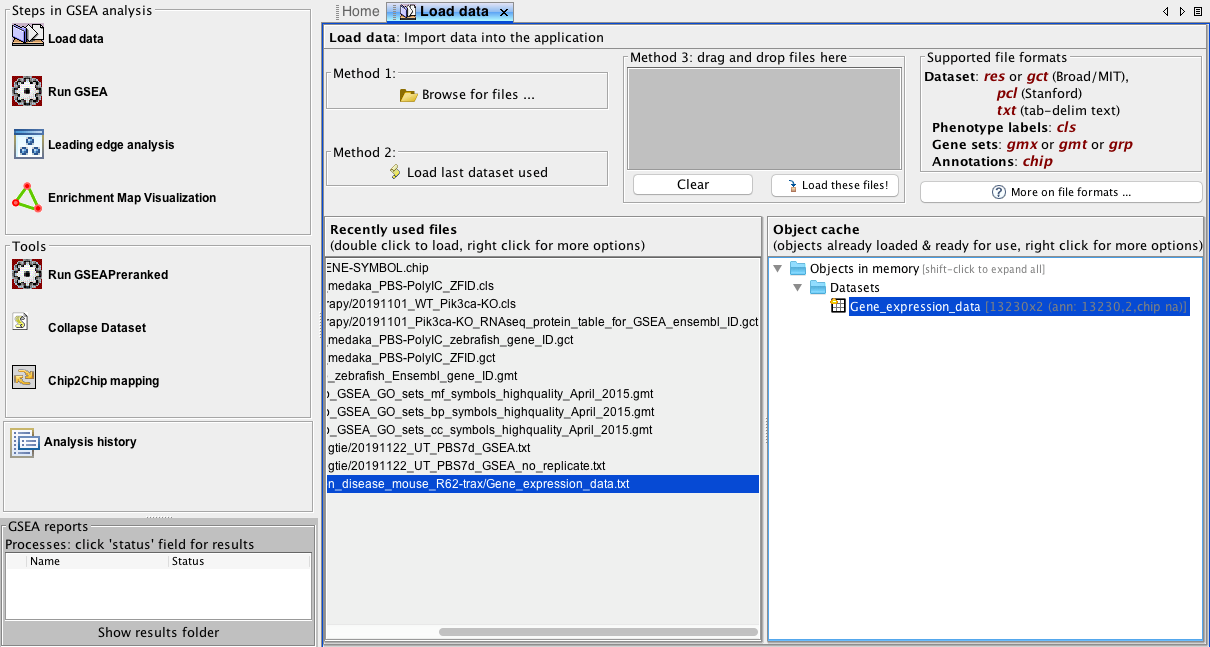
軟體安裝完成後點選圖示就可以直接執行,畫面如圖三。
圖三、GSEA軟體執行後的畫面,左邊是功能列表,中間是工作區域。
三、準備輸入資料
在圖一中的右邊圖示中我們可以看到GSEA分析需要兩種資料:基因表現資料(gene expression profile)與基因群組資料(gene set database)。其中後者在GSEA軟體已經提供相當豐富的資料(MSigDB),所以我們只要準備好基因表現資料即可。
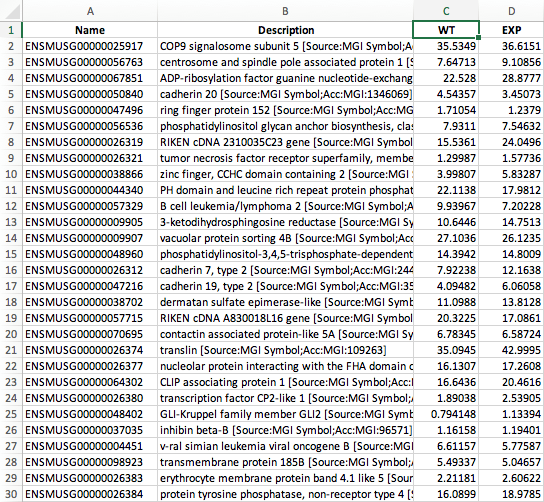
不過GSEA軟體僅支援某些固定的輸入格式,其中最簡單的是tab隔開的.txt (Tab delimited text) 文字格式檔案。這種格式我們可以利用Microsoft Excel來協助產生(如圖四)。
圖四、基因表現資料表範例。其中第一和第二欄是必要的。第一欄標題固定為Name,內容可以是Gene ID或是Gene Name等。第二欄標題固定為Description ,內容是對於基因的描述,內容沒有限制 (沒有的話可以補上N/A)。第三欄之後標題與欄位的數量沒有規範(應至少有控制與實驗組兩欄),內容則是樣本名稱與表現量數據。
編輯好之後存檔請選用「Tab間隔的文字檔案」(Tab delimited text .txt),如圖五。
圖五、Excel存檔時記得要選擇.txt檔案格式
四、匯入資料
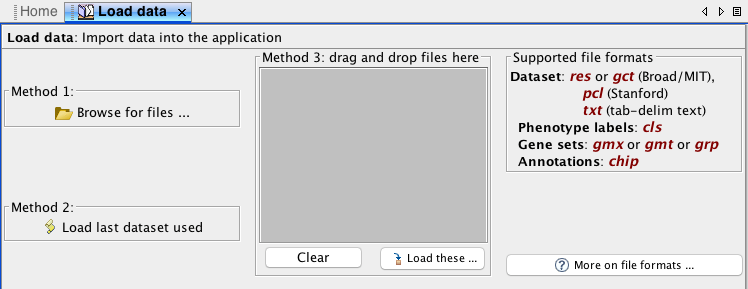
資料檔案準備好之後,就可以回到程式畫面(圖三)選擇左邊的Load data按鈕,出現如圖六的畫面後請在選擇走邊的Method 1: Browse for files… 來選擇剛剛準備好的資料檔案。
圖六、準備匯入檔案(Load data)的畫面
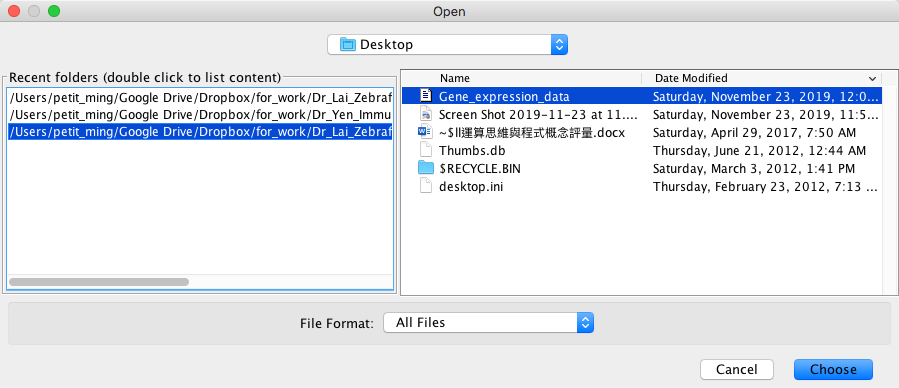
請從視窗中選擇要輸入的資料檔案(如圖七)。
圖七、選擇檔案視窗
輸入格式經過檢查正確之後出現資料已匯入畫面,在右下視窗出現輸入資料的名稱(如圖八)
圖八、右下視窗為目前已匯入的資料
五、選擇Gene sets
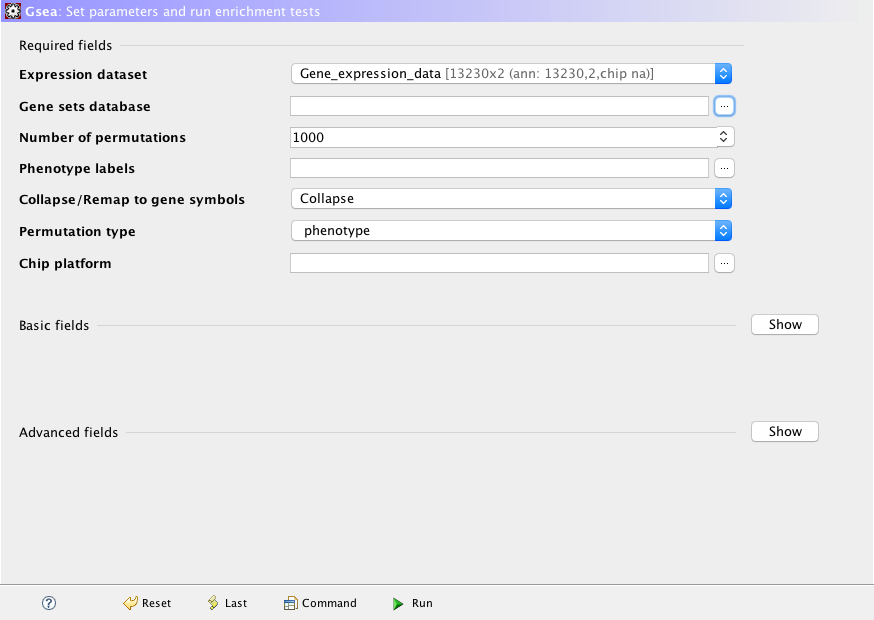
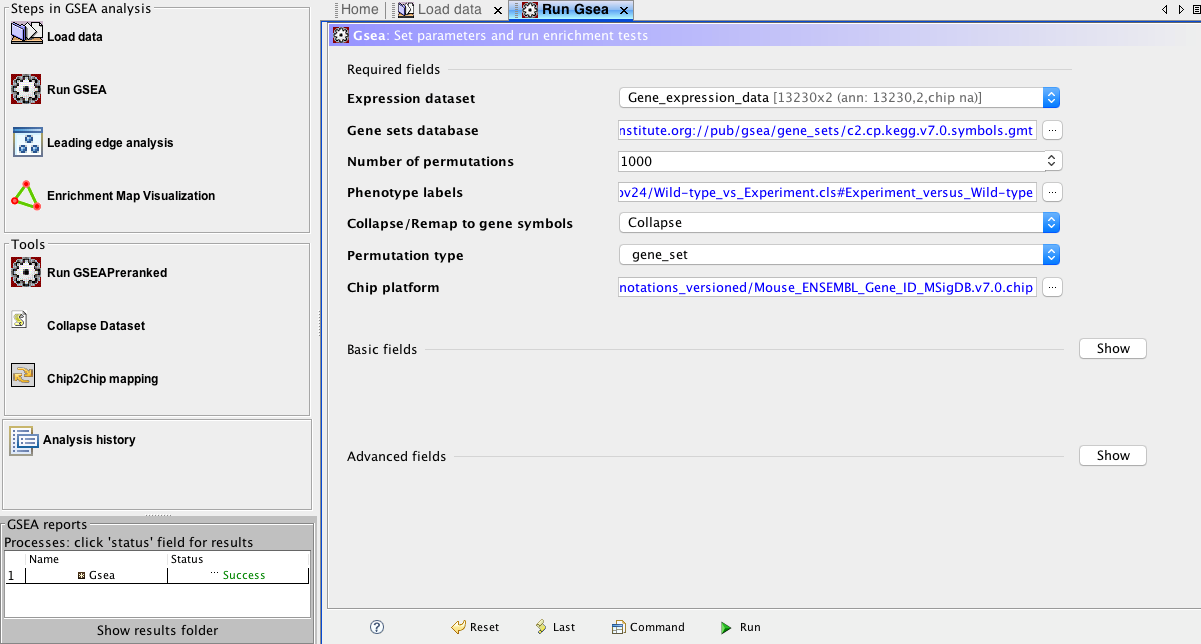
確認資料成功匯入後,我們先點選左邊的Run GSEA (圖八)功能。然後會出現設定分析參數的視窗(圖九)。我們由上而下一個一個來進行選擇設定。首先Expression dataset就是我們剛剛匯入的資料檔案。
圖九、參數設定視窗
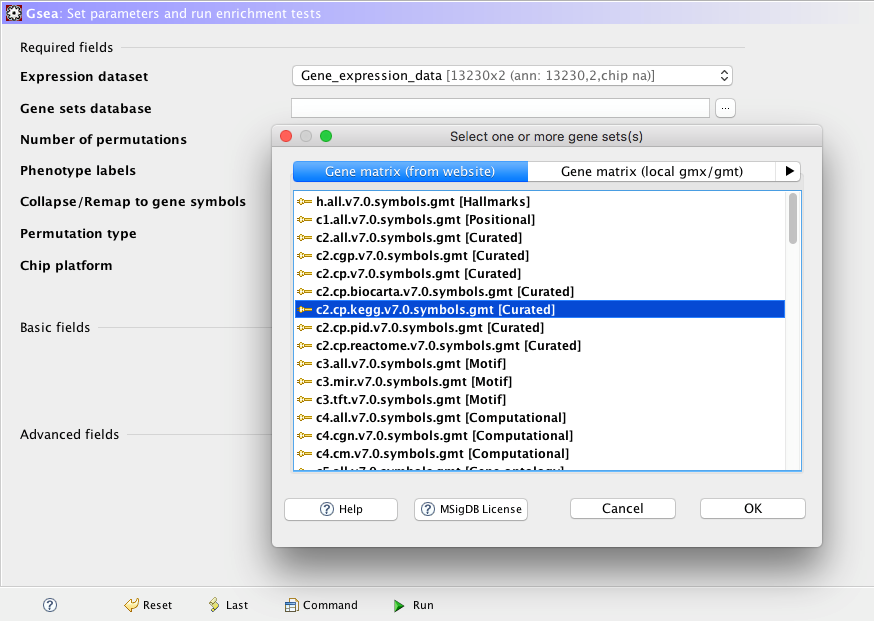
接下來我們要決定哪一個基因群來進行分析。先前有提到,在選單中GSEA已經提供了很多組基因群。一般來說我們會從KEGG pathway或是Gene Ontology的三種 categories, BP, CC, MF開始。這裡我們選擇KEGG當作範例(圖十)。
圖十、選擇要進行分析的基因群
六、設定比對群組
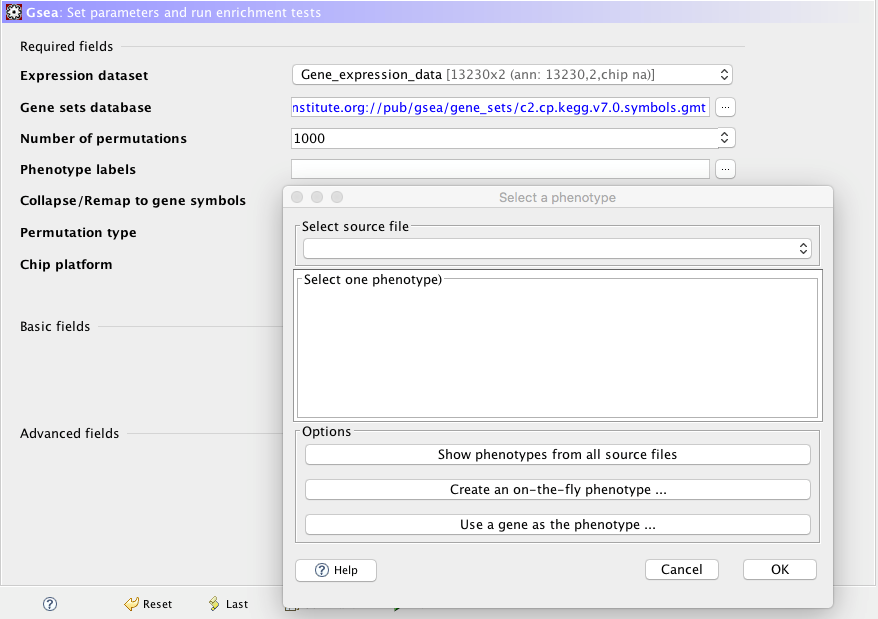
最重要的兩個輸入資料都設定好了之後,接者我們要告知城市哪些欄位的資料屬於控制組和實驗組(control class or experiment class)。點選Phenotype labels之後會出現下面的視窗(圖十一)。
圖十一、決定資料欄位的類別(控制組或是實驗組)
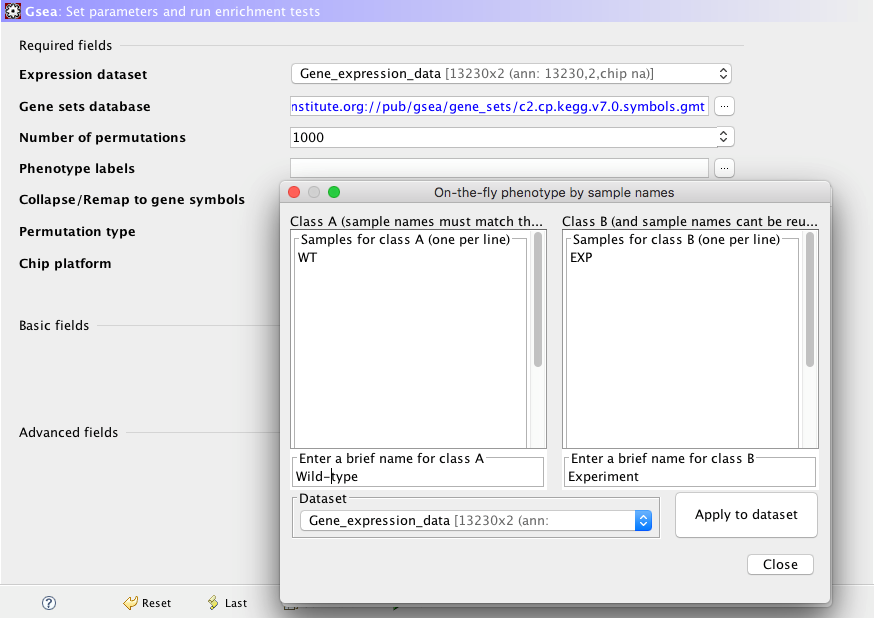
我們這裡點選 Create an on-the-fly phenotype … 的選項。
圖十二、設定欄位資料的類別視窗
由於我們的資料只有兩個欄位所以就依照原始輸入資料的欄位標題名稱(圖四)的填上去,並且在下方輸入兩個類別的名稱(如圖十二)。如果我們的資料包含有biological replicates的話,就可以在這裡指定哪些欄位同屬於一個類別。指定好之後點選右下角Apply to dataset即可。
七、選擇對應表
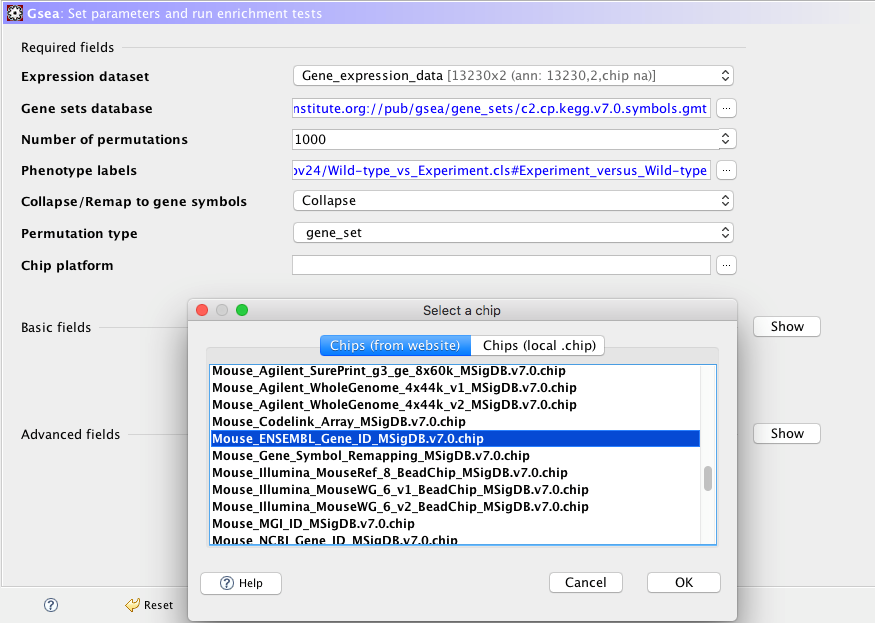
接下來在Collapse/Remap to gene symbols的選項維持預設的Collapse即可。但是在Permutation type的選項中因為我們的控制組會是實驗組都僅有一筆資料,所以這裏要選擇gene_set選項。但是如果我們的資料類別各有三個重複(replicates)或以上的話,則建議選擇phenotype選項。
最後一步是要決定輸入資料之間對應的方式,因為我們匯入的基因表現資料是以mouse Ensembl ID當做第一欄,所以這裡我們也要選擇相對應的資料轉換方式(如圖十三) ,若是非人類、小鼠或大鼠等物種在網路上也有不少編輯好的資料轉換檔案http://ge-lab.org/gskb/可供下載匯入使用)。
圖十三、選擇輸入資料之間的對應方式
八、進行分析
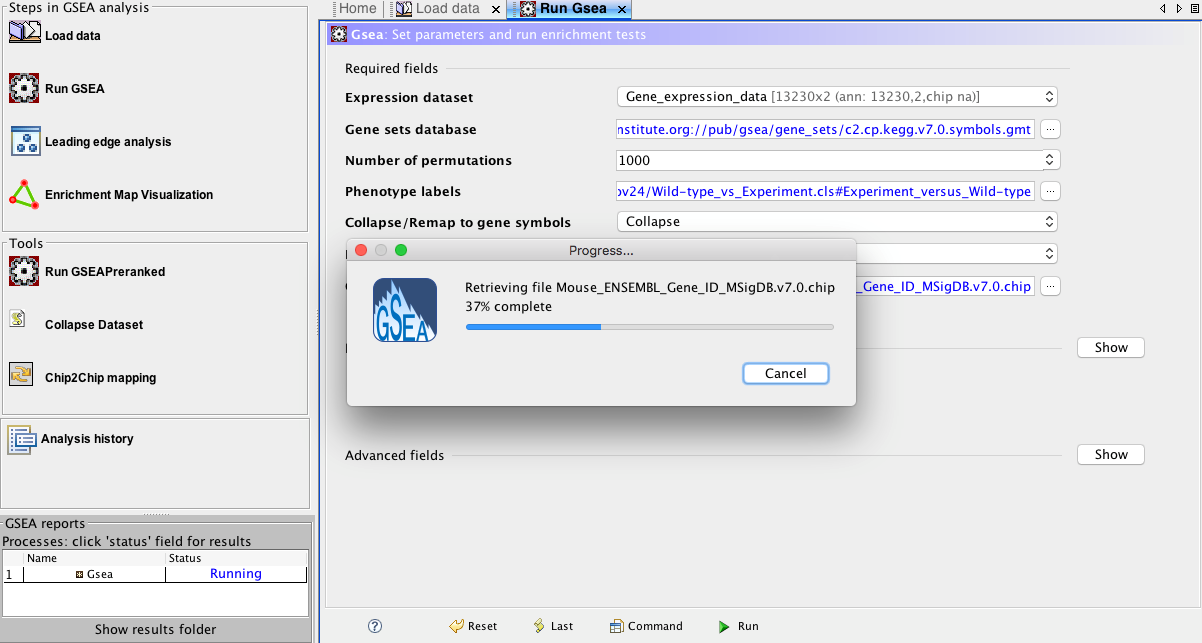
確認所有參數設定都完成了之後,要進行分析的話就是點選畫面最下面標注有Run字樣的綠色三角形符號即可。此時在畫面左下角會出現分析狀態為Running (藍色字體)如圖十四。
圖十四、程式正在下載資料與進行分析
分析完成後,原來的畫面左下方的藍色字體Running狀態會改為綠色字體的Success。接者請點選該綠色字體的Success,程式會自動開啟一個瀏覽器視窗,並把所有的結果顯示在結果網頁中了(圖十六)。
圖十五、分析完成
圖十六、結果報告的網頁