圖文/ 洪慈懋(創源生物科技股份有限公司分子數位處)
本次的IPACase Study,將會介紹IPA知識庫所累積及整合的資料來源,輕鬆的幫祝您了解研究中關鍵的目標分子和疾病適應症關係,希望藉由此案例探討,能幫助大家更了解系統生物學的奧妙。
•建立目標分子連接到tumor microenvironment (TME)和疾病的網絡
•剖析癌症中的關鍵免疫分子
•分析TME途徑內表達變化的影響
•建立與免疫反應有關的監管概況
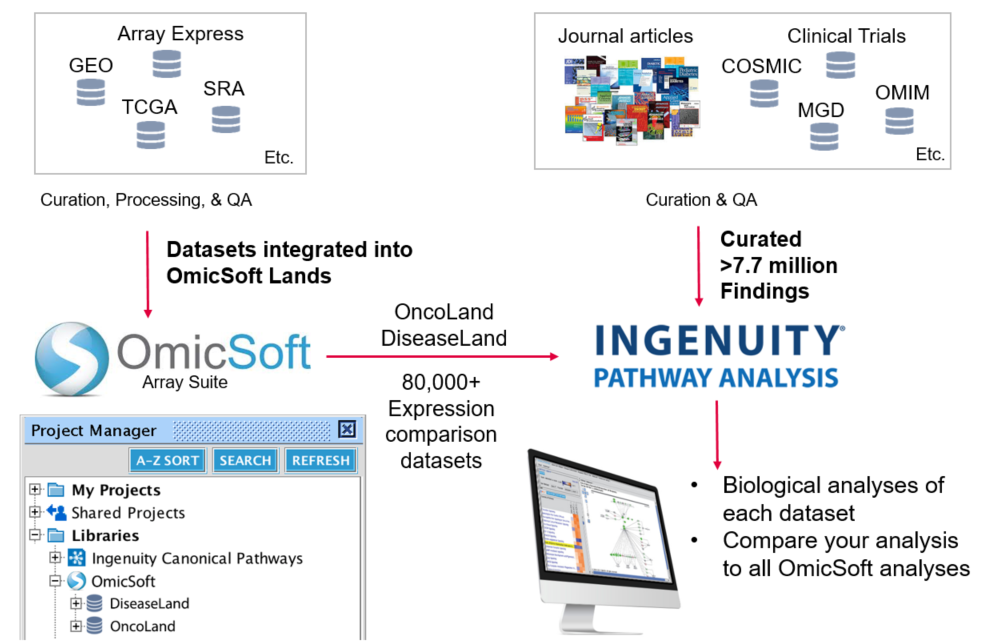
QIAGEN Knowledge Base and OmicSoft Lands
QIAGEN知識庫目前經過專業人員校正及跟自動化比對的資料,及整合了OmicSoft中八萬筆以上的疾病、致癌知表現資料,也有第三方資料庫如 COSMIC(癌症) 、Target Scan(miRNA)、OMIM等,周周更新累積20年有7.7 百萬資料,也因為有這麼龐大的資料庫,使用戶們可以很好的利用IPA找尋出來關鍵的分子的關係。
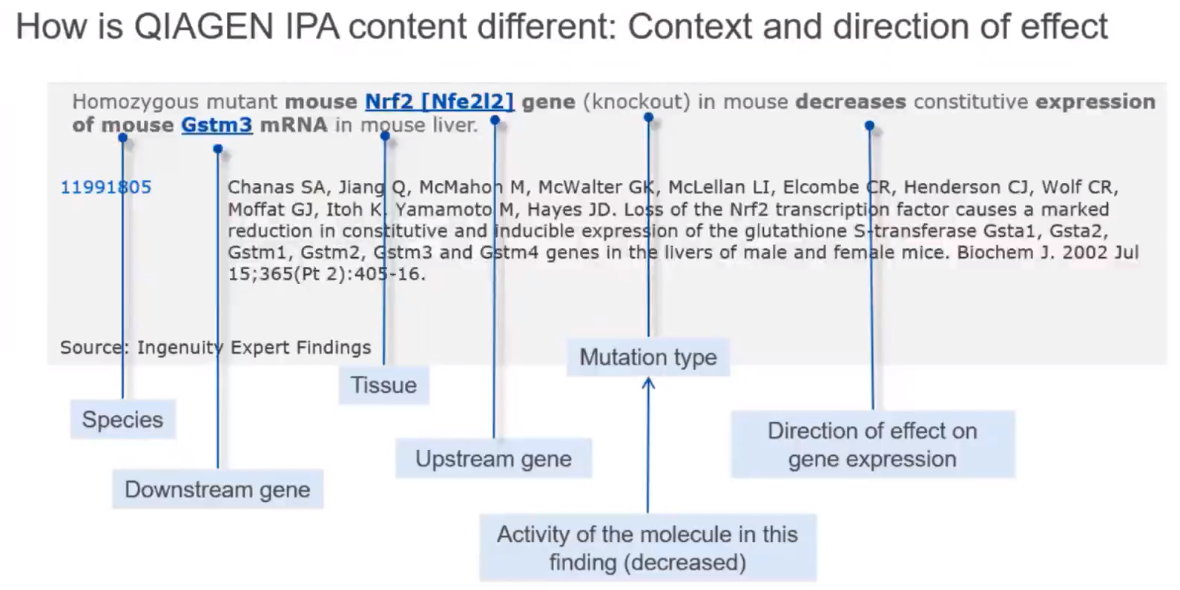
QIAGEN IPA 資料搜尋結構
人工校閱過的資料結構我們會重整為資料如上,資料整理上會細分為物種、組織、上游基因及表現方式等,並會以專業人士整理文獻以快速了解相關重點。
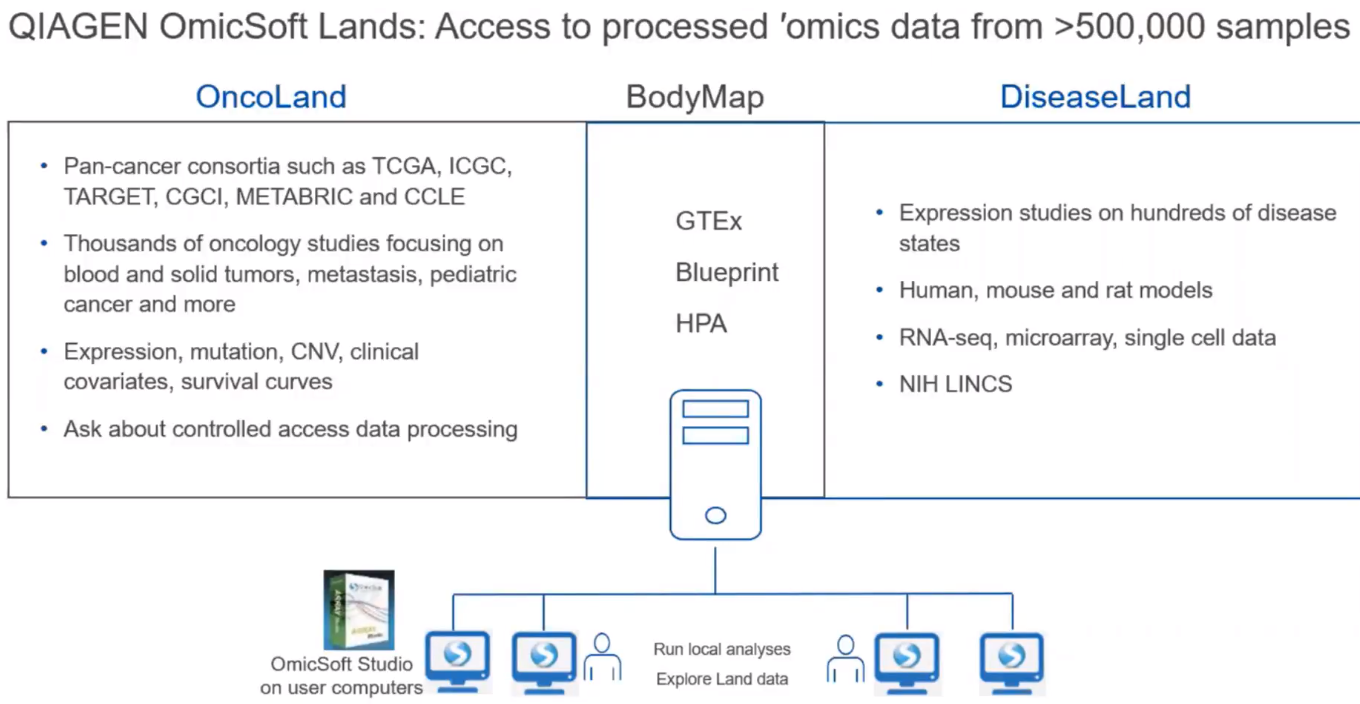
QIAGEN OmicSoft Lands
在IPA當中最重要的分子表現資料庫為OmicSoft lands,收錄資料則為萃取50萬筆 samples,這個資料庫會在分為兩大塊Oncoland & Diseaseland (內含資料參考圖),這些資料我們著重在了解metadata & description,並將其相互比對分析,最後這些資料我們會在BODYMAP上呈現其表現資訊。
Case Study: Investigating the tumor microenvironment
Voronov, Elena, and Ron N. Apte. "Targeting the tumor microenvironment by intervention in interleukin-1 biology." Current pharmaceutical design 23.32 (2017): 4893-4905.
此篇文獻是Interleukin 1, IL-1白血球介素1族包括11種細胞因子,它們組成了促炎細胞因子的複雜網絡。這些細胞因子通過調控白血球和內皮細胞的整合素的表達,啟動和控制炎症反應,也與TME的關係息息相關。
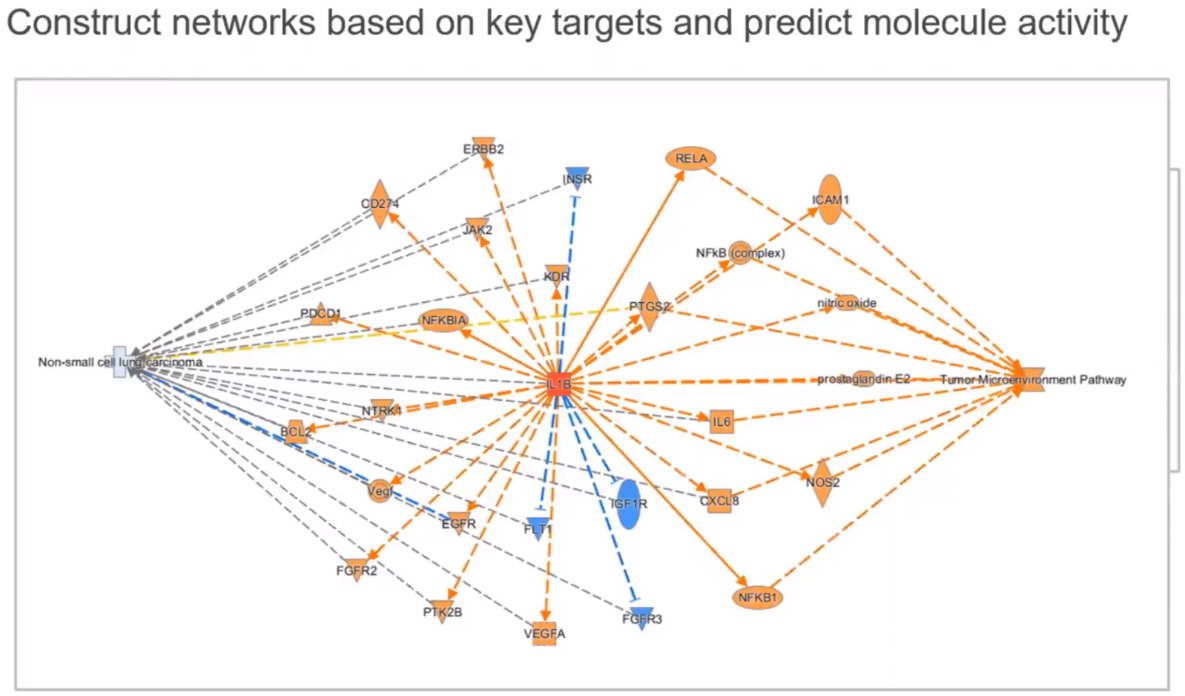
上圖表示了 IL1B, Non-small cell lung carcinoma(LC) & Tumor Microenvironment Pathway (TMP)的關聯性,我們也可以顏色來去評斷在IL1B表現上升時,針對pathway或其他的下游基因的表現評斷資訊(by reference),在這次的介紹,我們將會教大家如何建立出三者之間之交互作用網路。
Network建立 (Search – Build – Connect – Path Explore)
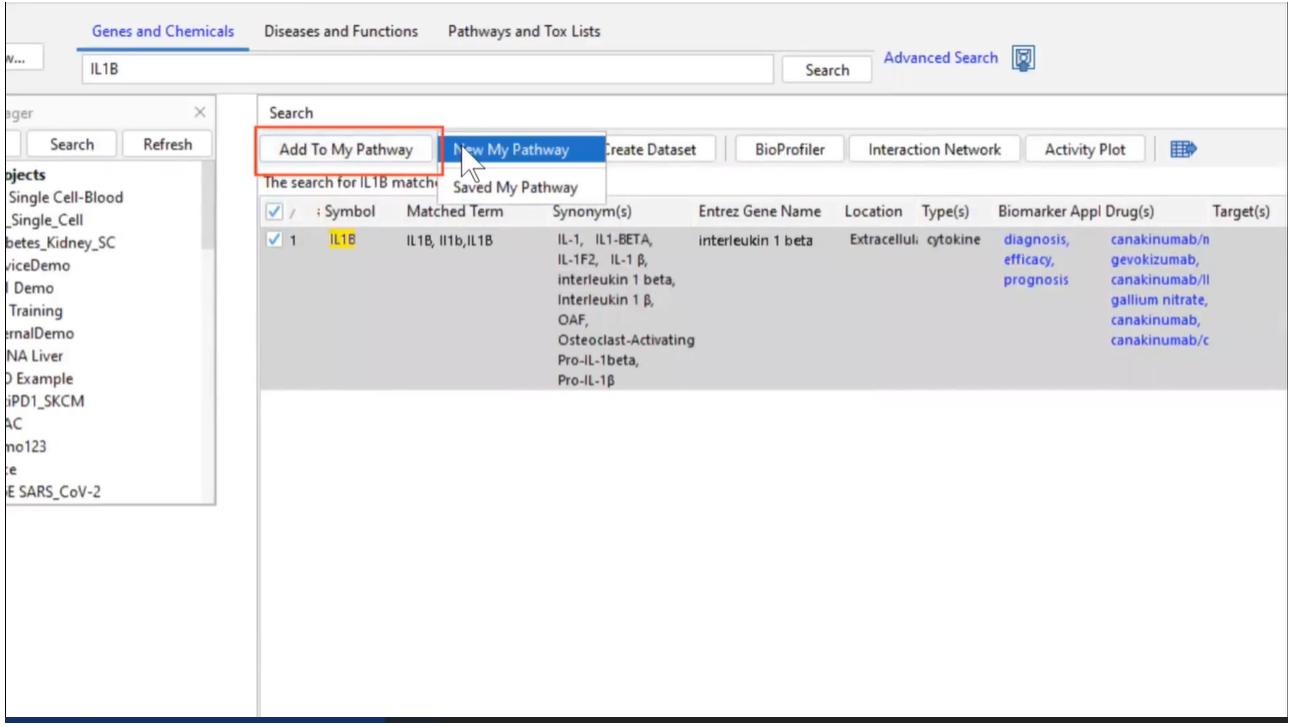
首先在IPA的軟體介面,上方欄位為資料的查詢頁面,分為基因及化合物、疾病及功能、生物路徑,我們在基因欄位,搜尋”IL1B”,目標物勾選後,將其Add To My Pathway。
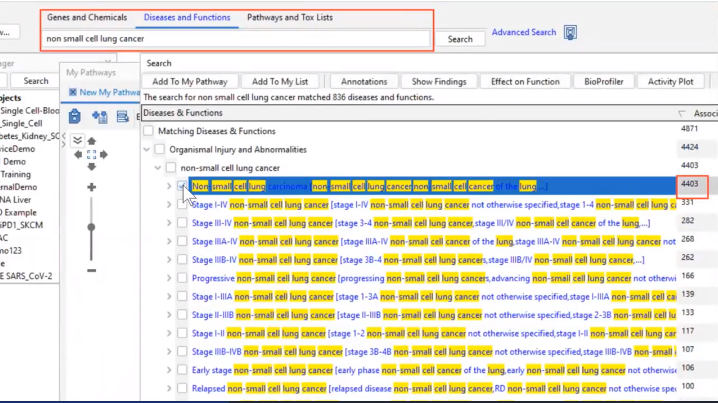
在疾病欄位,搜尋”non small lung cancer”,選擇最主要的疾病及相關分子最多者,也加入my pathway。
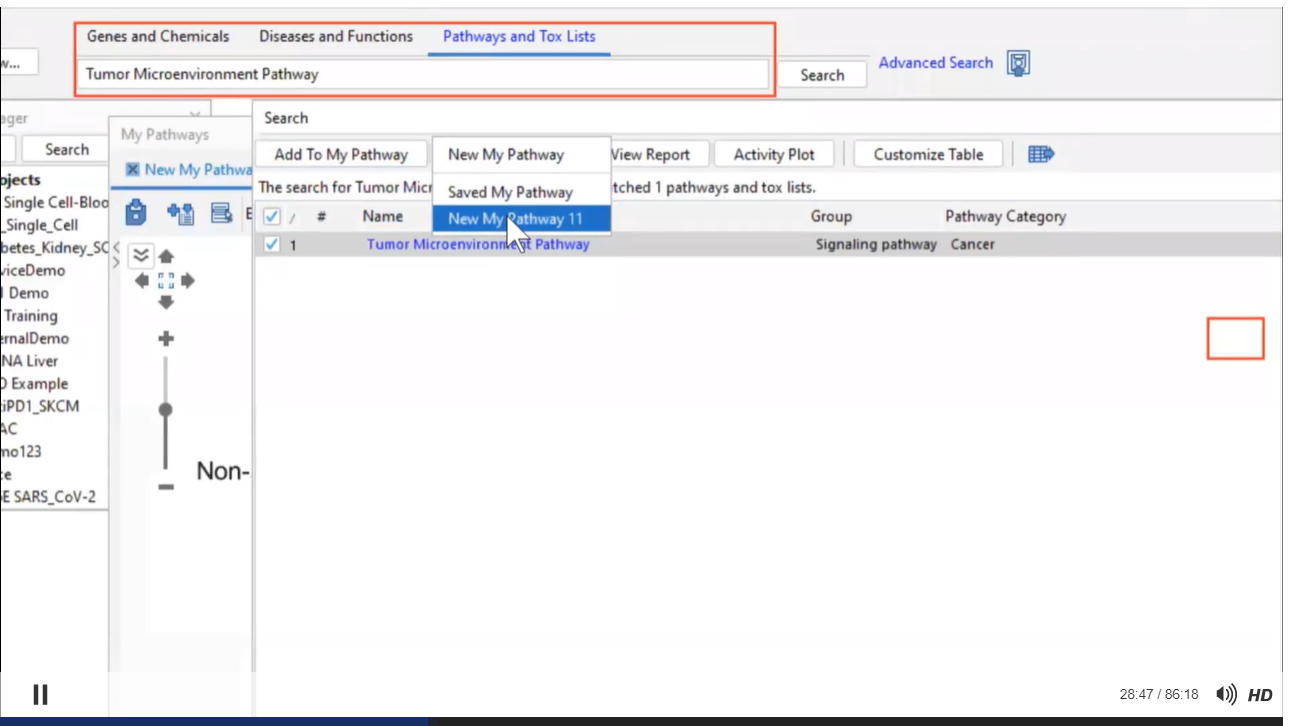
同樣的方法在路徑欄位,搜尋”Tumor Microenvironment Pathway”,將其加入my pathway。
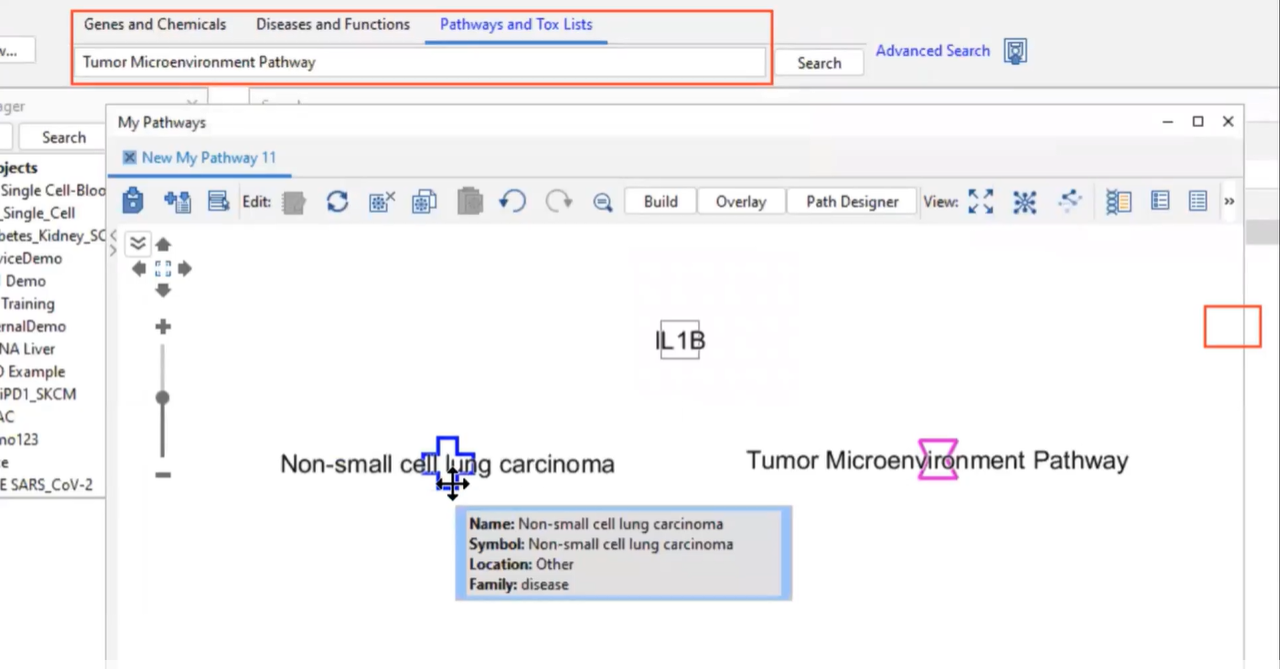
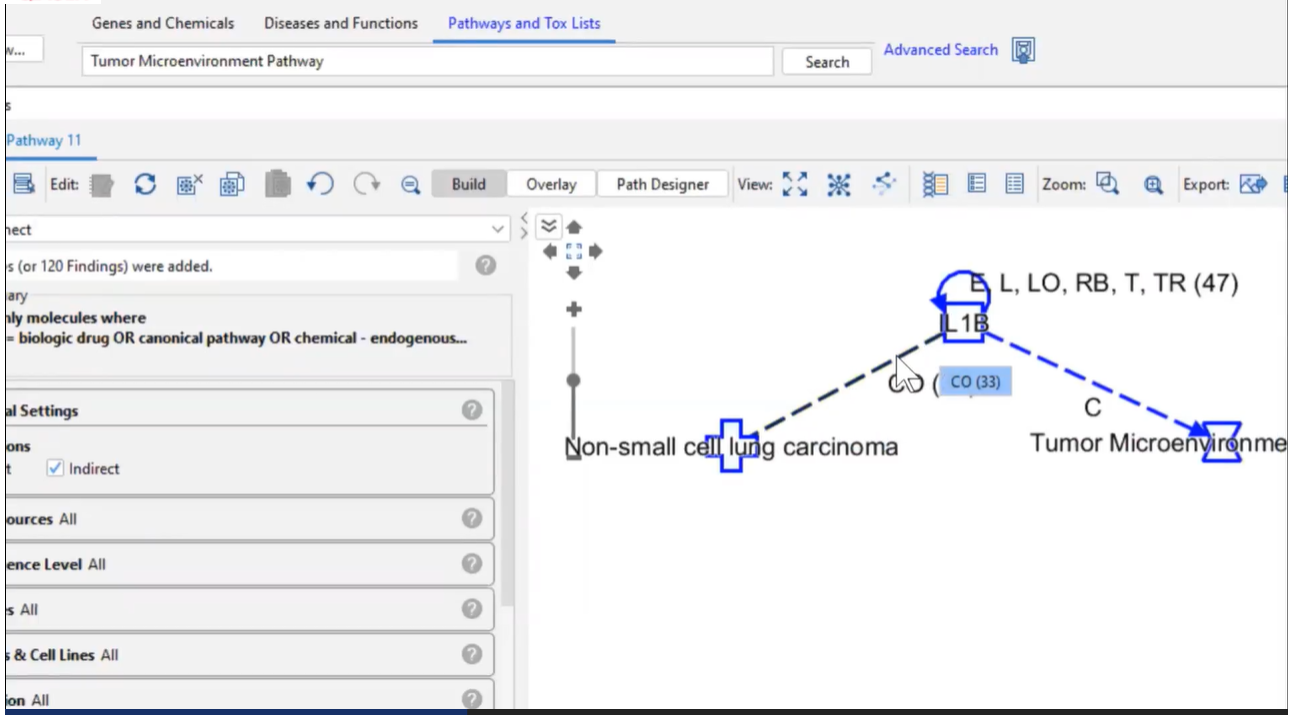
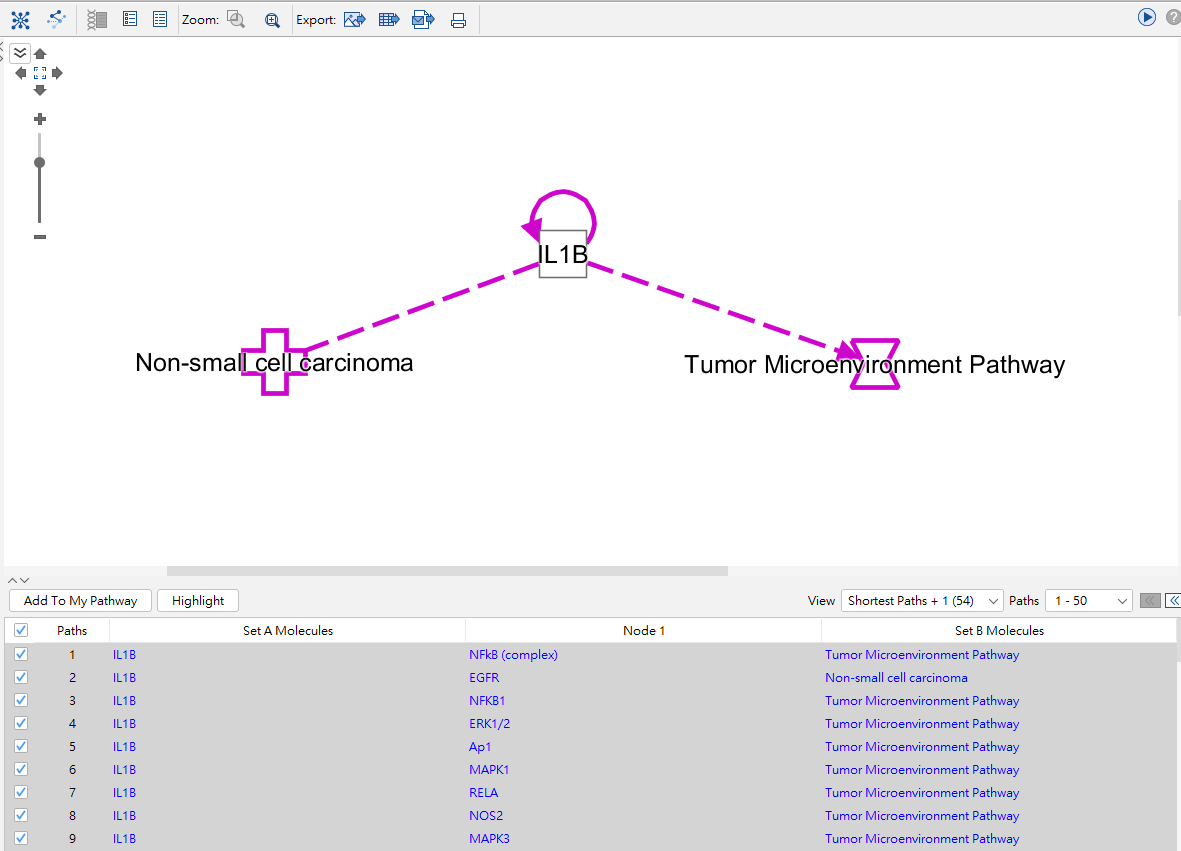
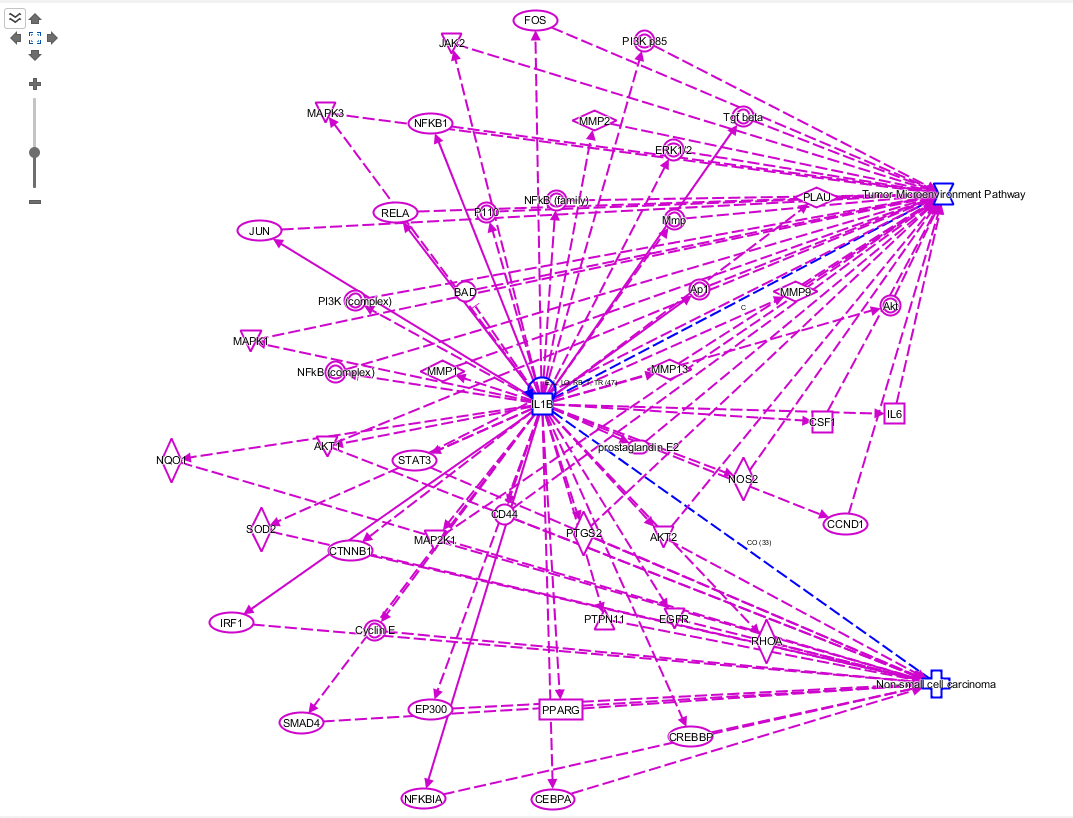
完成後將會產生如上圖的畫面,接著我們將會透過IPA的建立功能(Build)來進一步去探討三者之間更進一步的交互作用機制為何。
路徑圖中,點選Build功能,下拉工具選單選擇connect,可先用預設條件建構三者間關係。
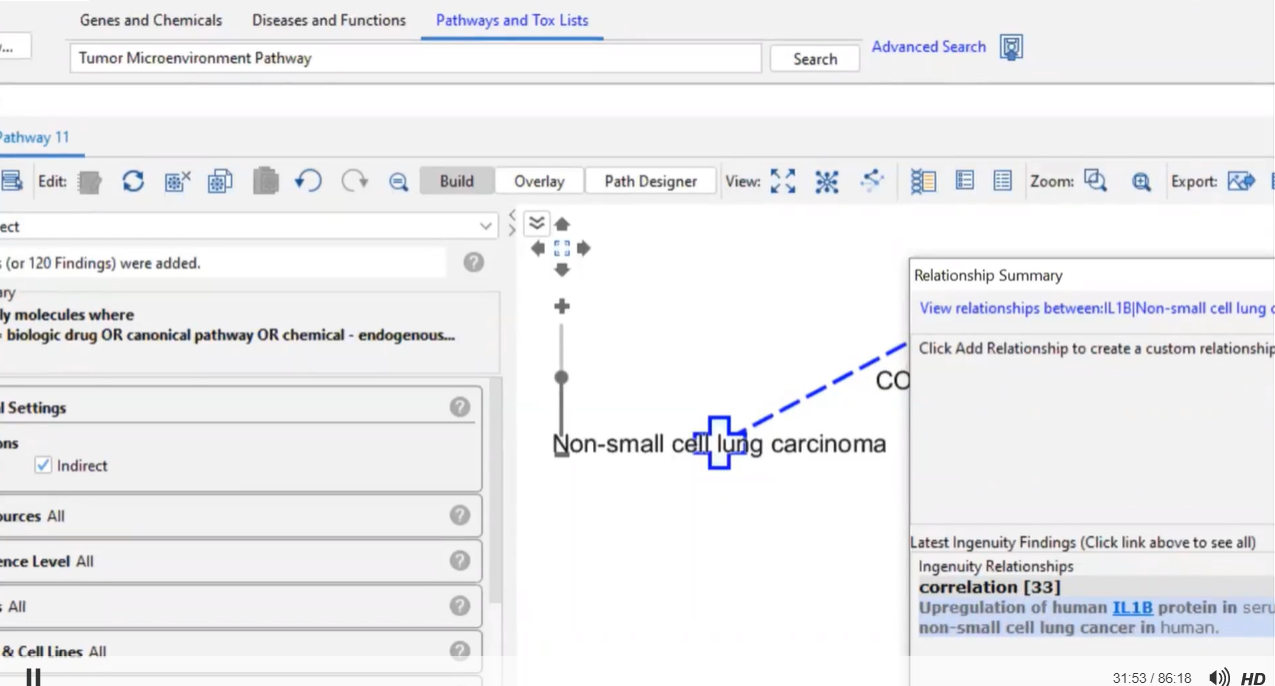
點選任兩分子之間的連結,可查詢分子之間的關係。
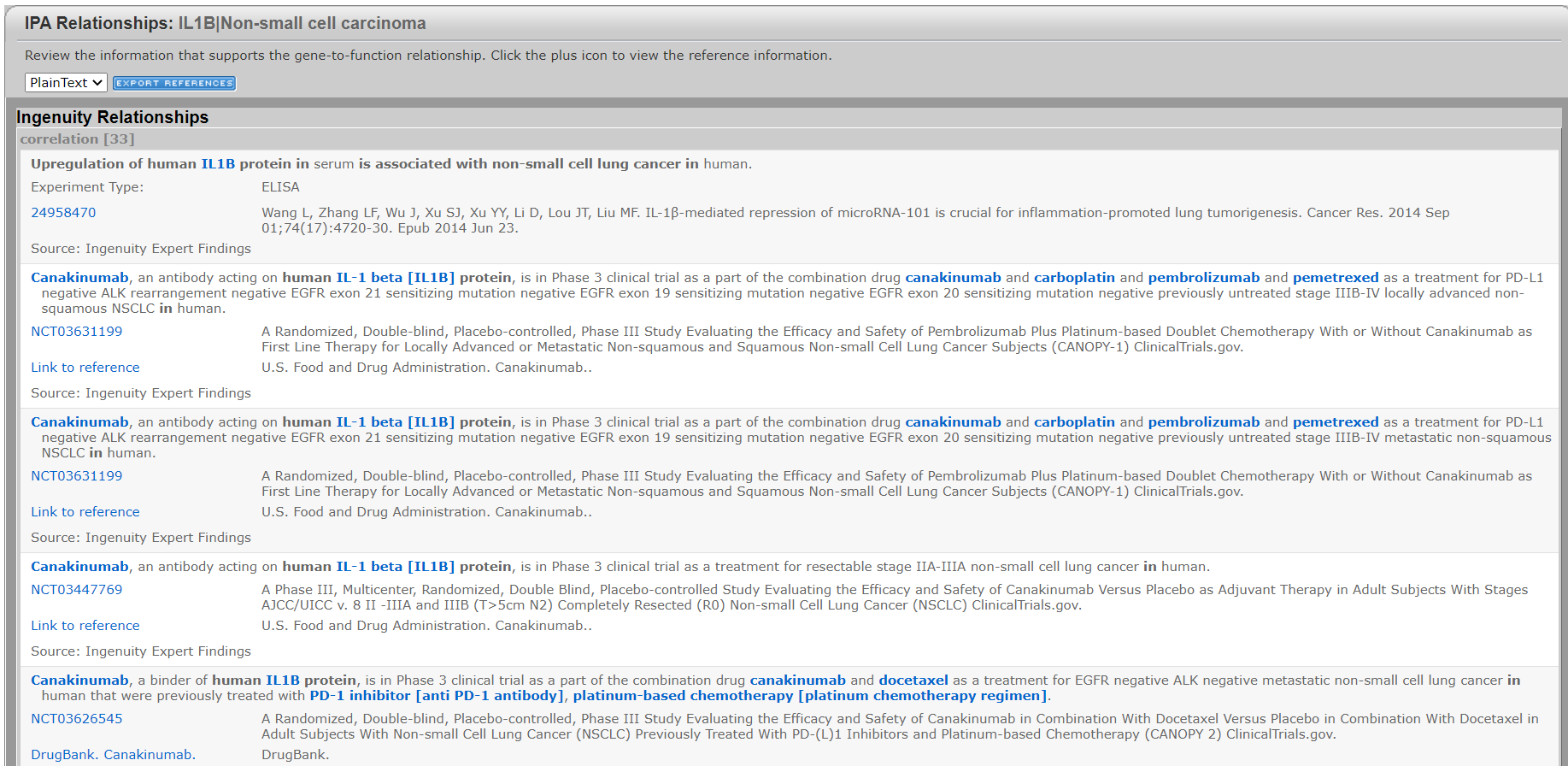
例如: IL1B及Non-small cell lung carcinoma的關係屬於CO(Correlation)目前有33篇文獻記載,接著再點選”view relationship”即可從網頁上查找所有記載的文獻資料。
將會以網頁的方式條列整理所有文獻資訊,並詳細記載文獻重點、來源、PubMed ID等,也可快速的export資訊供大家利用。
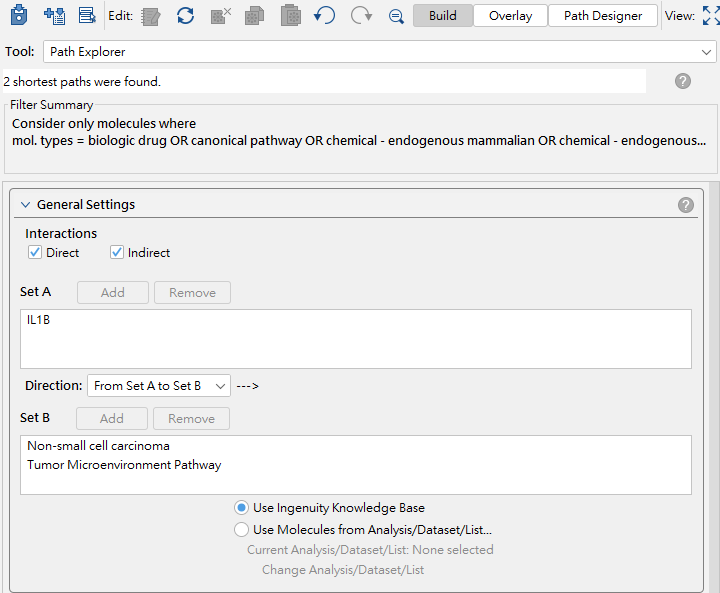
接著為了進一步探討IL1B與LC+TMP的關係,我們利用Path Explore的功能,將IL1B設定於Set A,LC+TMP設定於Set B,並指定其方向為”From Set A to Set B”。
我們在條件列,可於Relationship Type設定為: activation, causation, inhibition。在路徑計算後,在右下角View的位置設定最短路徑為+1,並將其全部勾選後新增。
以上便可以快速地利用IPA的”connect”、”Path Explore”功能,先找出IL1B, LC & TMP三者間關鍵的交互作用。
Network分析 (Overlay – MAP - )
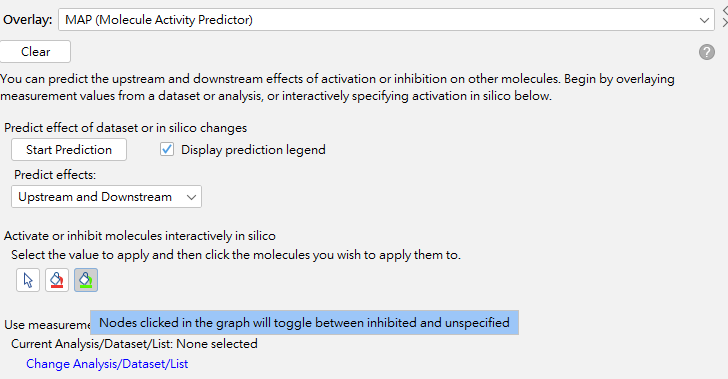
這部分的分析我們將利用IPA中的預測上下游的功能,及omicsoft資料庫比對來為大家做介紹。
首先點選Overlay功能,下拉工具選單選擇MAP(Molecule Activity Predictor),可先選擇紅色區塊,先將IL1B設定為activated代表,接著點選Start Prediction。
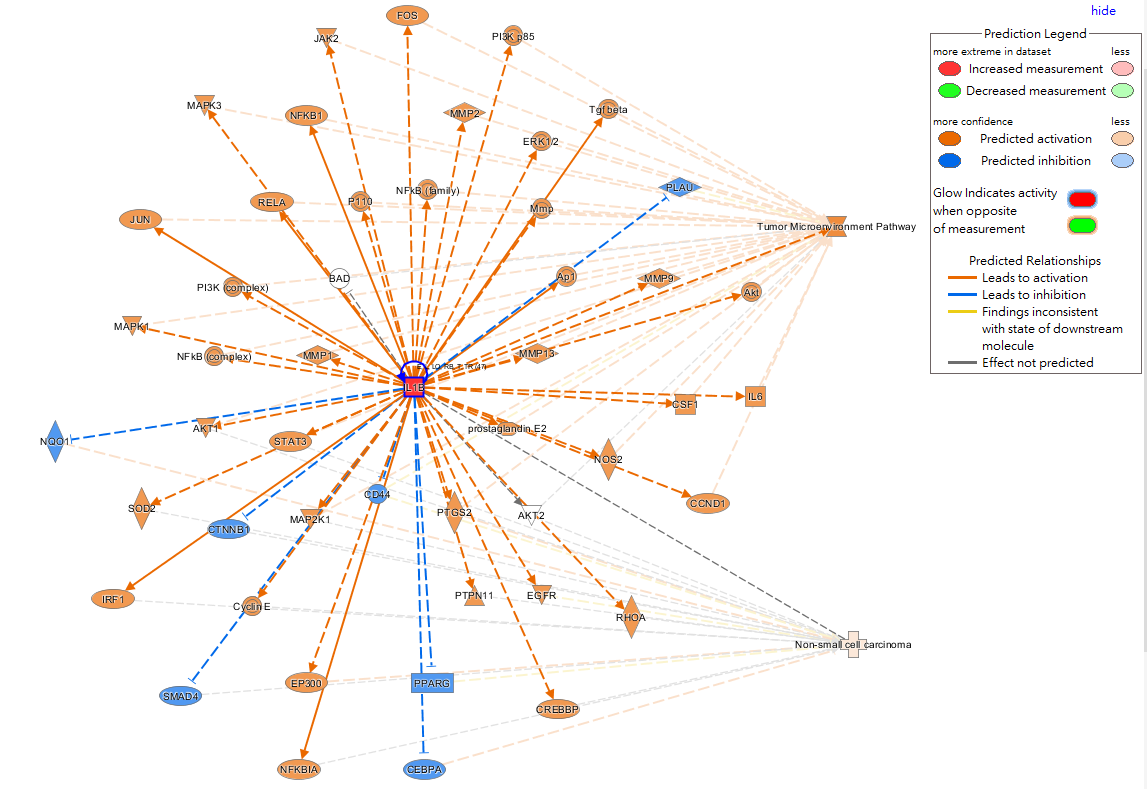
從上圖的MAP預測,可看到說當IL1B活化時,同時也會造成LC及TMP的活化。
除了IL1B直接影響TMP路徑,也可以發現IL1B會間接活化MMP1,在造成TMP的活化;同樣也能從網路中觀察到,IL1B間接活化PTGS2,進而造成LC及TMP的活化。
以上範例介紹,我們可先透過IPA的資料庫建立,先探討有興趣的分子、疾病及路徑,並利用”connect”、”Path Explore”、”MAP”等功能串接IL1B, LC & TMP三者間關鍵的交互作用。
-
如何利用IPA資料庫搜尋,快速搜尋各分子之資料,及兩兩之間關係,並快速整理現有文獻資訊。
-
利用關鍵目標分子建立交互作用網路,並透過計算預測方式了解各分子間,是如何去影響網路內的成員。
這期的案例探討,我們熟悉了生路網路建置的基本功能後,在下一期的案例探討,將會繼續帶大家比對分析實驗數據,及透過Omicsoft資料庫(In Analysis Match Function)查找更多的資訊。
更多詳細資料可參考: Link
生圖購買的IPA是同時上線2人,因此須採取預約時段使用。申請註冊及預約說明請詳見IPA網頁。歡迎多多利用。