圖文/ 洪慈懋(創源生物科技股份有限公司分子數位處)
本次的IPA Case Study,一樣會接續應用IPA知識庫所累積及整合的資料來源,輕鬆的幫助您了解研究中關鍵的目標分子和疾病適應症關係,將延續上次的範例,教大家如何套用真實數據做分析比較,能幫助大家更深入系統生物學的應用。
•建立目標分子連接到tumor microenvironment (TME)和疾病的網絡
•剖析癌症中的關鍵免疫分子
•分析TME途徑內表達變化的影響
•建立與免疫反應有關的監管概況
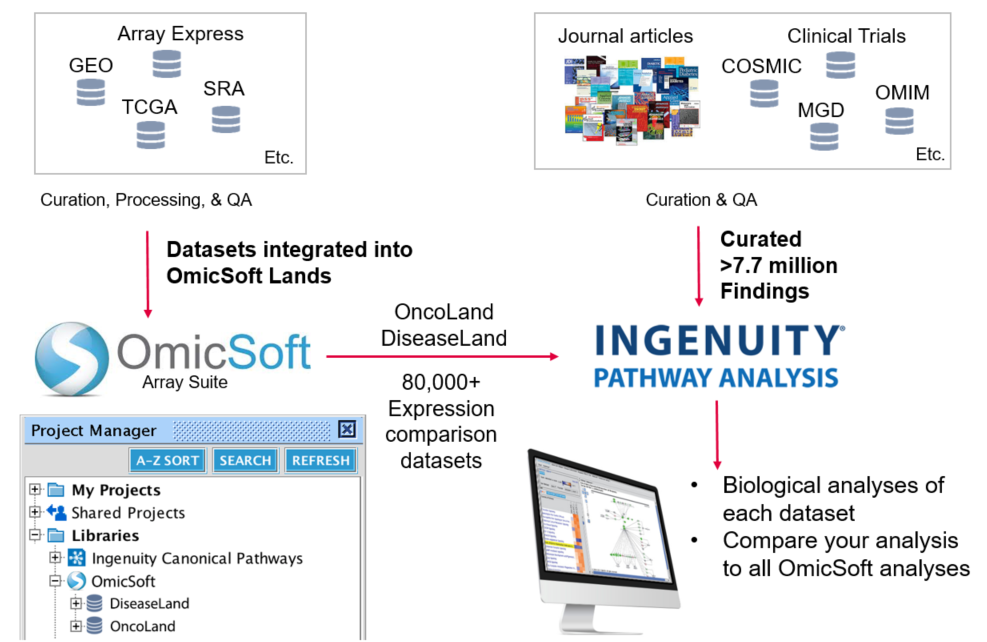
QIAGEN Knowledge Base and OmicSoft Lands
QIAGEN知識庫目前經過專業人員校正及跟自動化比對的資料,及整合了OmicSoft中八萬筆以上的疾病、致癌知表現資料,也有第三方資料庫如 COSMIC(癌症) 、Target Scan(miRNA)、OMIM等,周周更新累積20年有7.7 百萬資料,也因為有這麼龐大的資料庫,使用戶們可以很好的利用IPA找尋出來關鍵的分子的關係。
Case Study: Investigating the tumor microenvironment
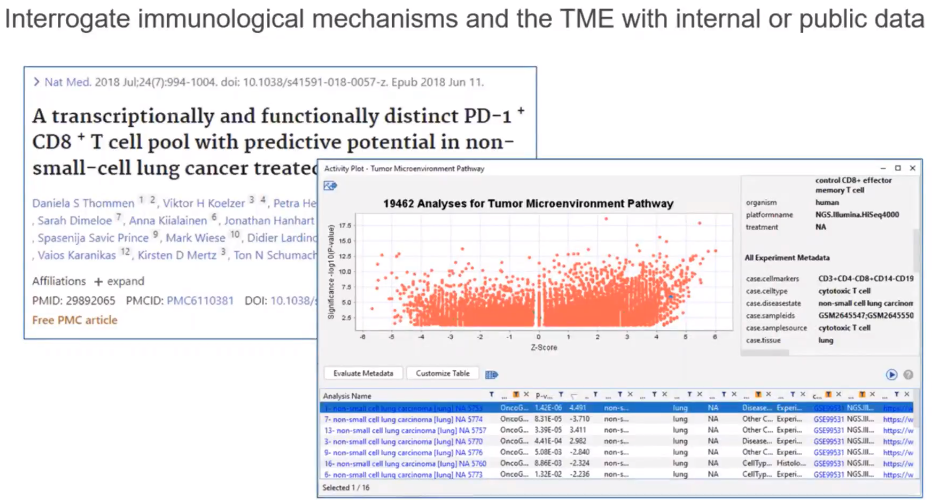
THOMMEN, Daniela S., et al. A transcriptionally and functionally distinct PD-1+ CD8+ T cell pool with predictive potential in non-small-cell lung cancer treated with PD-1 blockade. Nature medicine, 2018, 24.7: 994-1004.
上次案例我們分享了如何找出 IL1B, Non-small cell lung carcinoma(LC) & Tumor Microenvironment Pathway (TMP)之間的關聯性,而這次的案例將分享如何透過QIAGEN知識庫及公開資料來探討免疫機制及腫瘤微環境(TME)之關係,並比較各個不同非小細胞癌的病例之間的表現變化及分析。
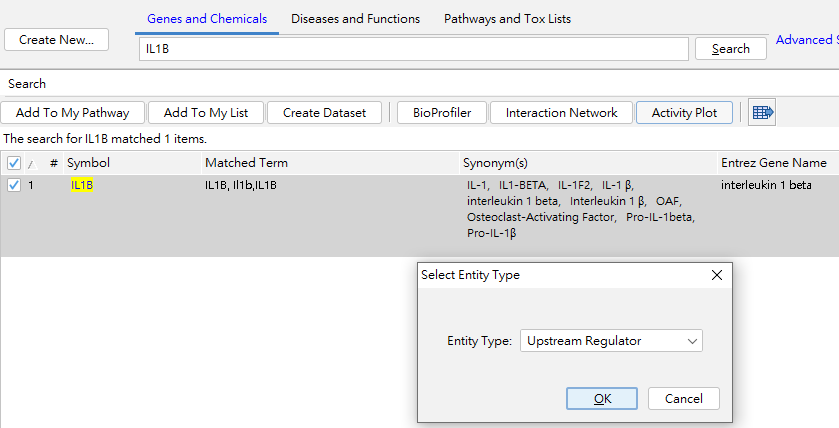
資料建立 (Search – Activity Plot)
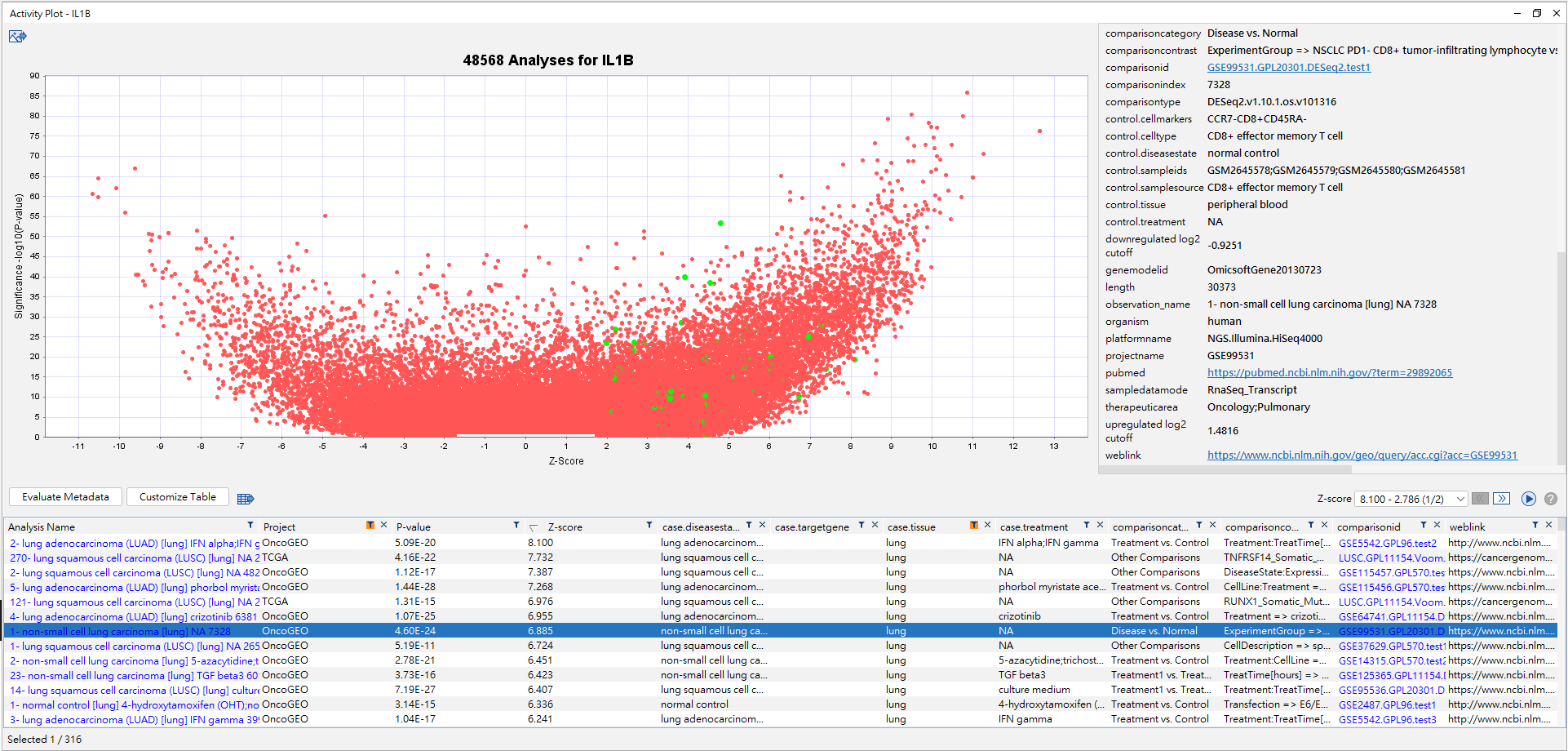
首先在IPA的軟體介面,基因欄位搜尋”IL1B”,目標物勾選後,點選Activity Plot,Type選擇Upstream Regulator.
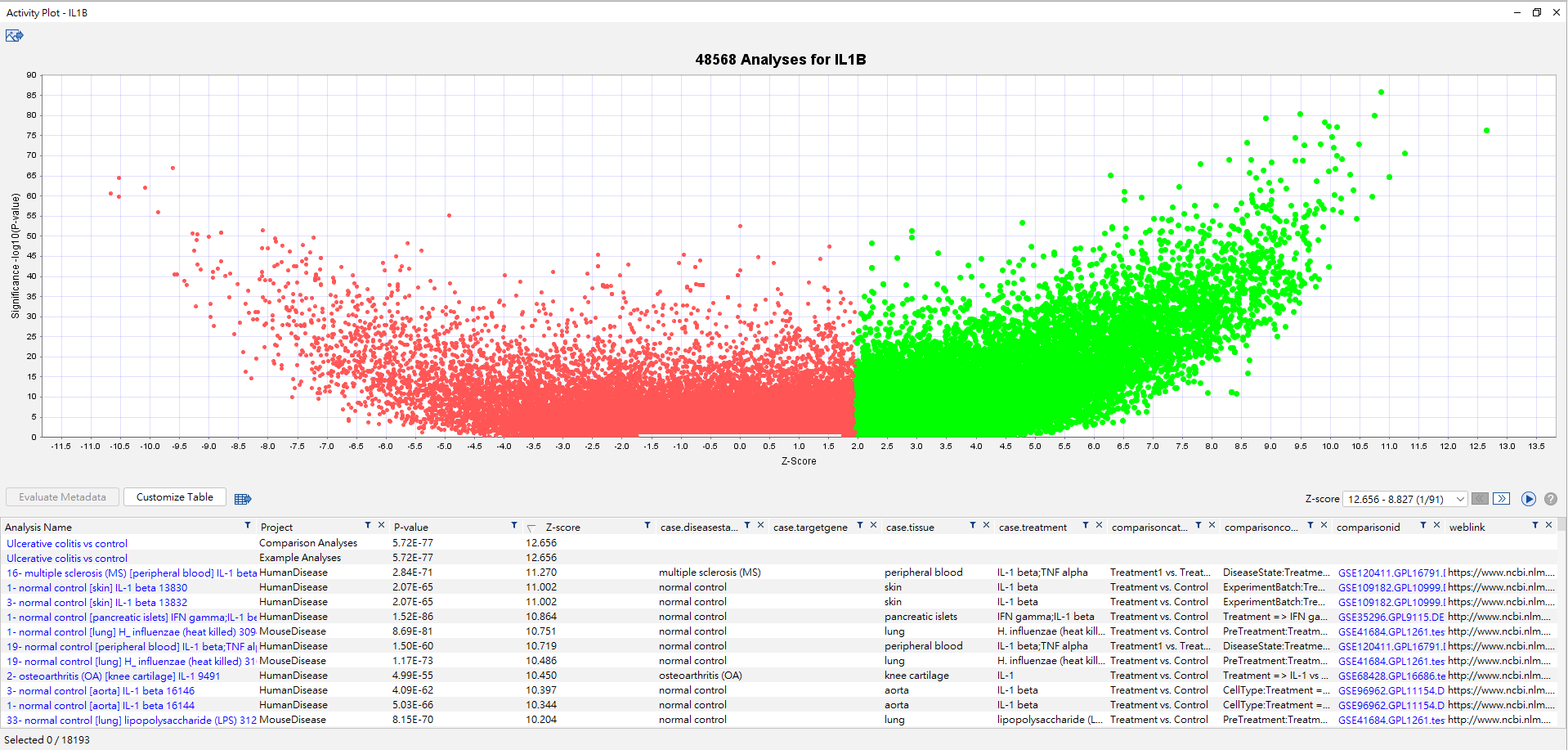
搜尋後的結果,可得到IL1B約五萬篇分析資料有表現資料,我們可以先將Z-Score大於2的資料集框起來(上圖綠色處),可得到各筆資料之名稱、計畫、P-value、Z-score、組織、疾病狀態…等相關資訊。
進一步,我們針對Project: OncoLand及case.tissue: *lung* 做資料篩選,可得到約300筆相關之分析資料,點選任一個有興趣的analysis name,右上方會出現此筆資料之數據注釋(metadata),包含了細胞型態、疾病狀況、組織、疾病及正常態的比較資訊,以及所有實驗資料之細節。(metadata之最下方為weblink,可自動連結至NCBI的網站,查找有興趣的文獻實驗,如下圖。)
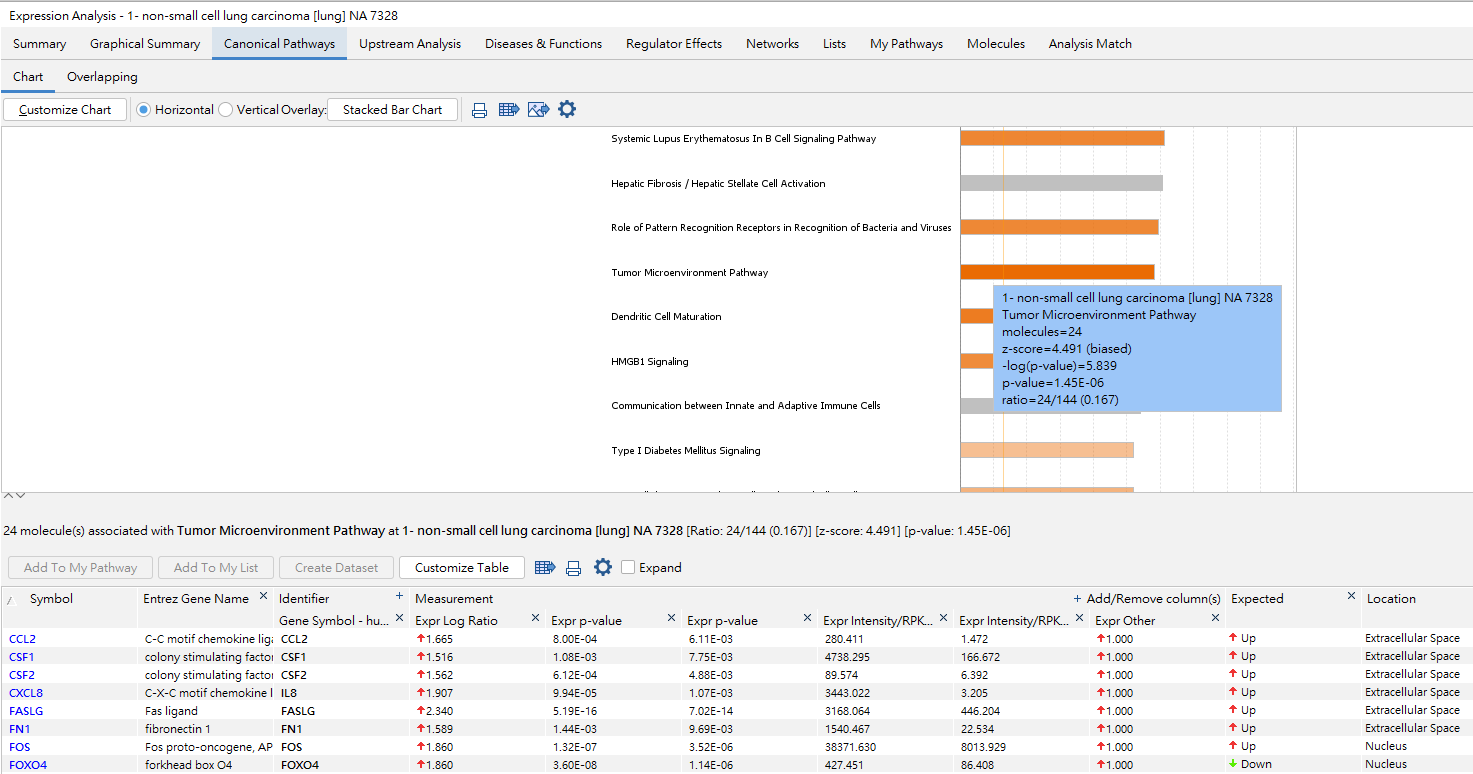
接著我們將帶大家分析TME途徑內表達變化的影響,先點選1-non-small cell lung carcinoma[lung] NA7328的IPA分析資料,可得到已透過IPA核心分析的GSE99531資訊,包含圖形、路徑、上游調控因子、疾病功能、調控、分子網路及資訊比對。
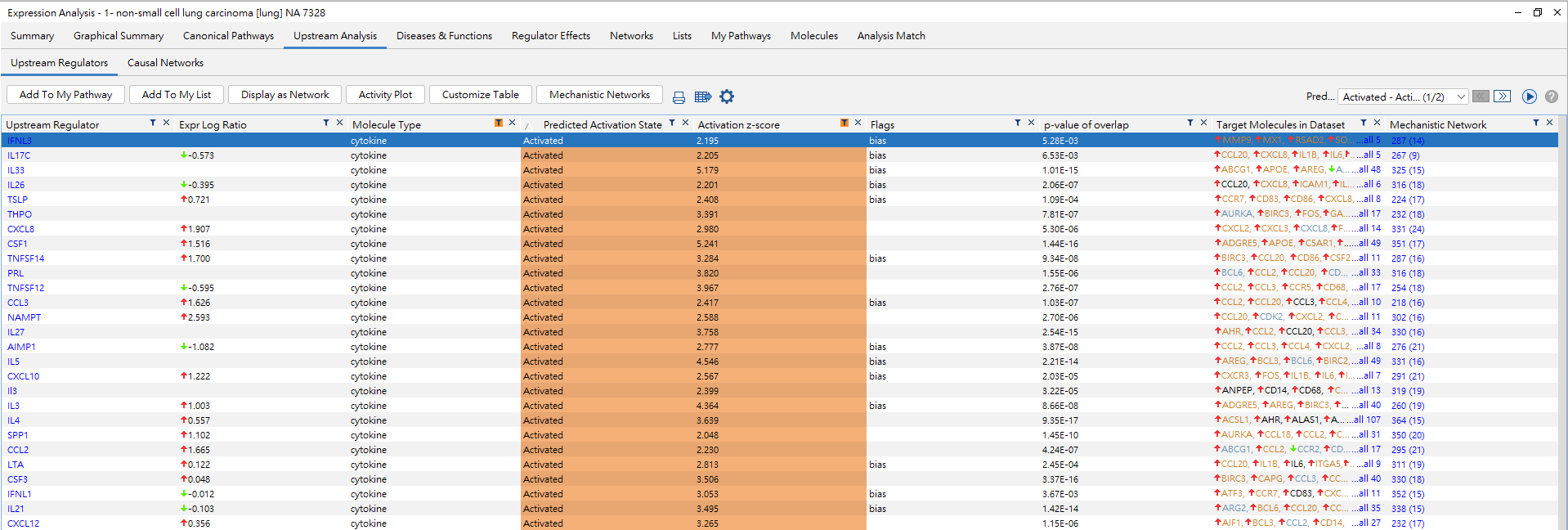
點選”Canonical Pathways”,可得到IPA找出所有相關(p-value)及影響(z-score)之生物路徑,選擇我們今天的主題TME pathway,在NA 7328中共有24個分子參與,Z-score: 4.491預測此路徑為活化表現。
下方的詳細資料則會顯示完整24個分子的資訊,包含名稱、實驗數據、預測表現、位置。
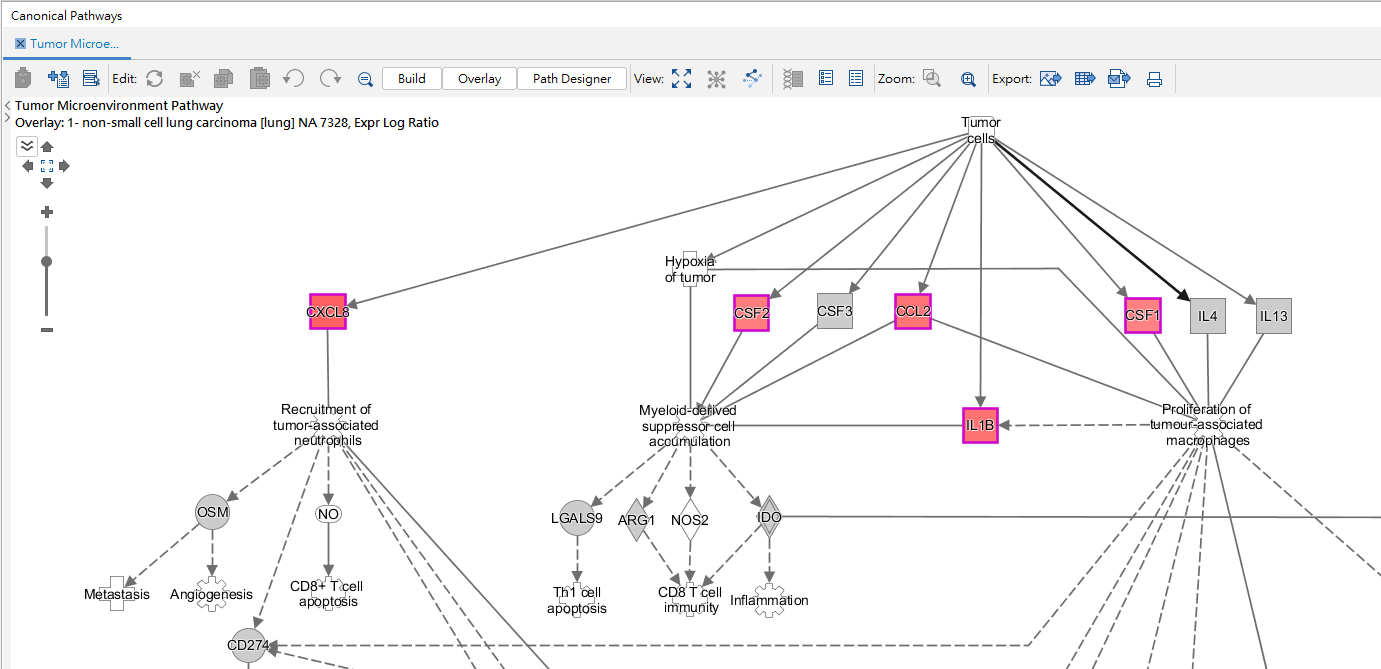
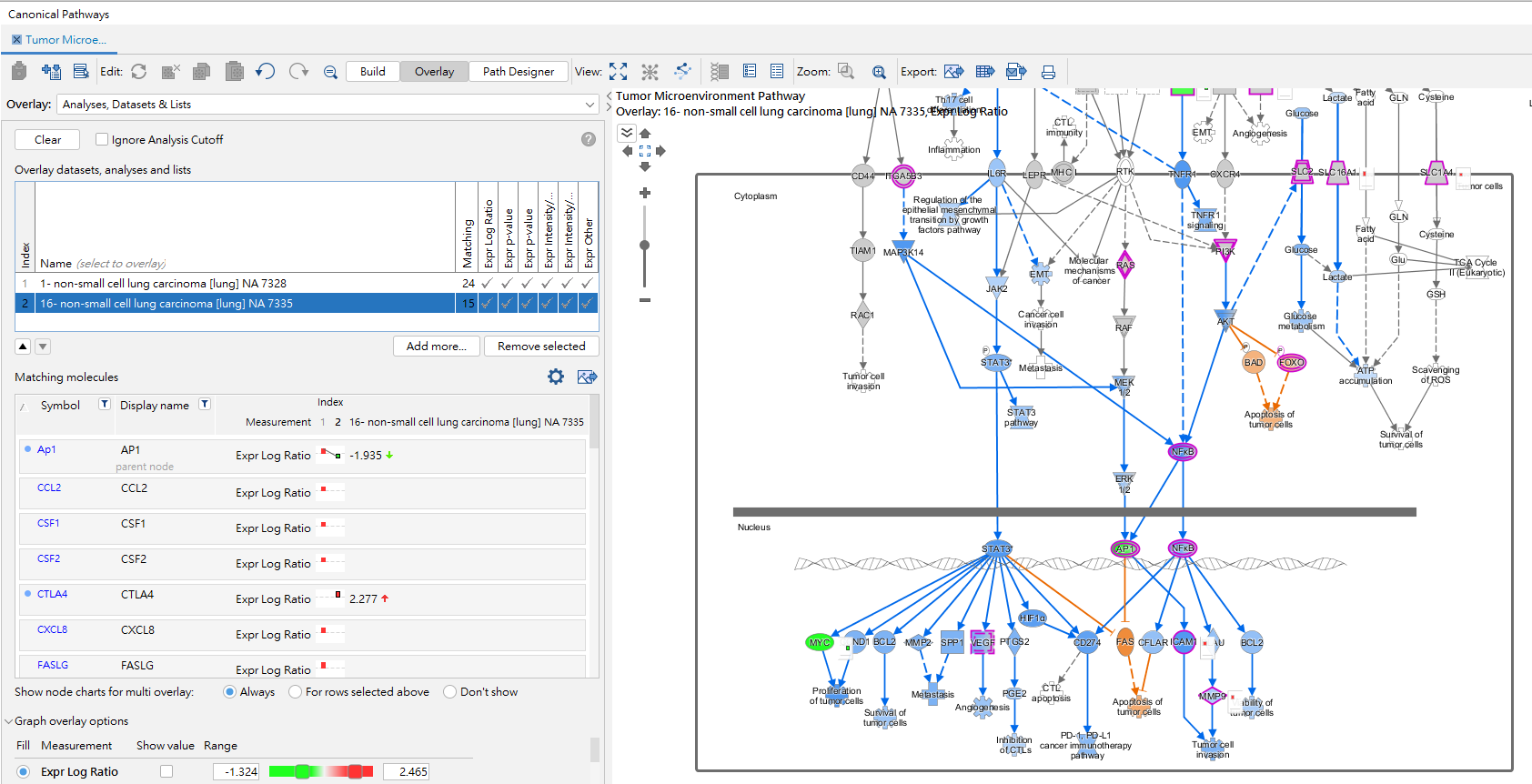
點開TME路徑圖,圖中有顏色的分子表示此份文獻有影響的,紅色的為高表現,綠色的為低表現,我們可以發現上游的Tumor cells可影響到CXCL8、CSF1、CSF2、CCL2、IL1B分子,進而導致(leads to)與腫瘤反應、增殖(recruitment、proliferation)等功能。
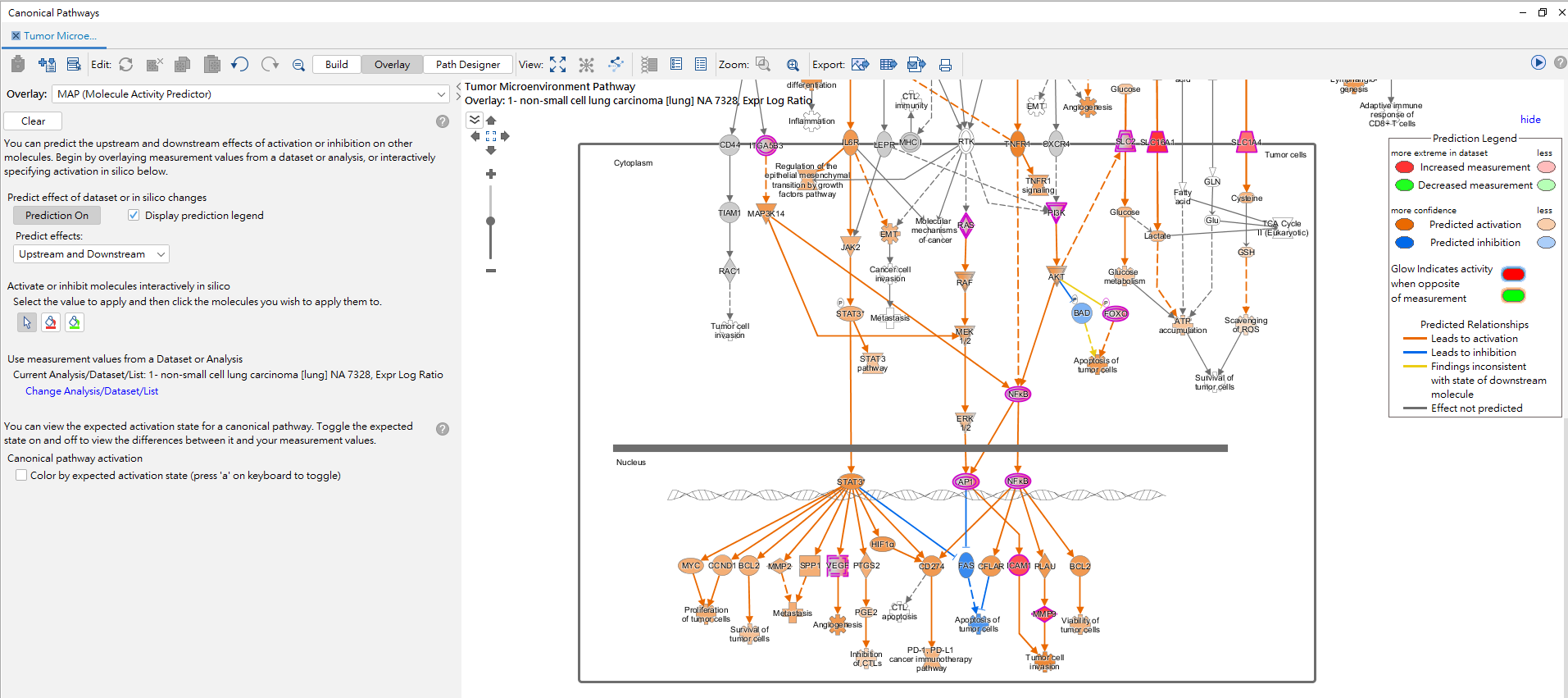
另外我們也可以使用功能(overlay -> MAP),透過IPA快速預測在TMP下游腫瘤細胞的影響,可看到於細胞質活化SLC1A4、SCLC16A1、PI3K、NFkB等分子,進而活化了細胞核中STAT3、MYC、BCL2…等分子,並增加了腫瘤細胞的proliferation、survival及apoptosis相關表現及PD-1, PD-L1 cancer immunotherapy pathway.
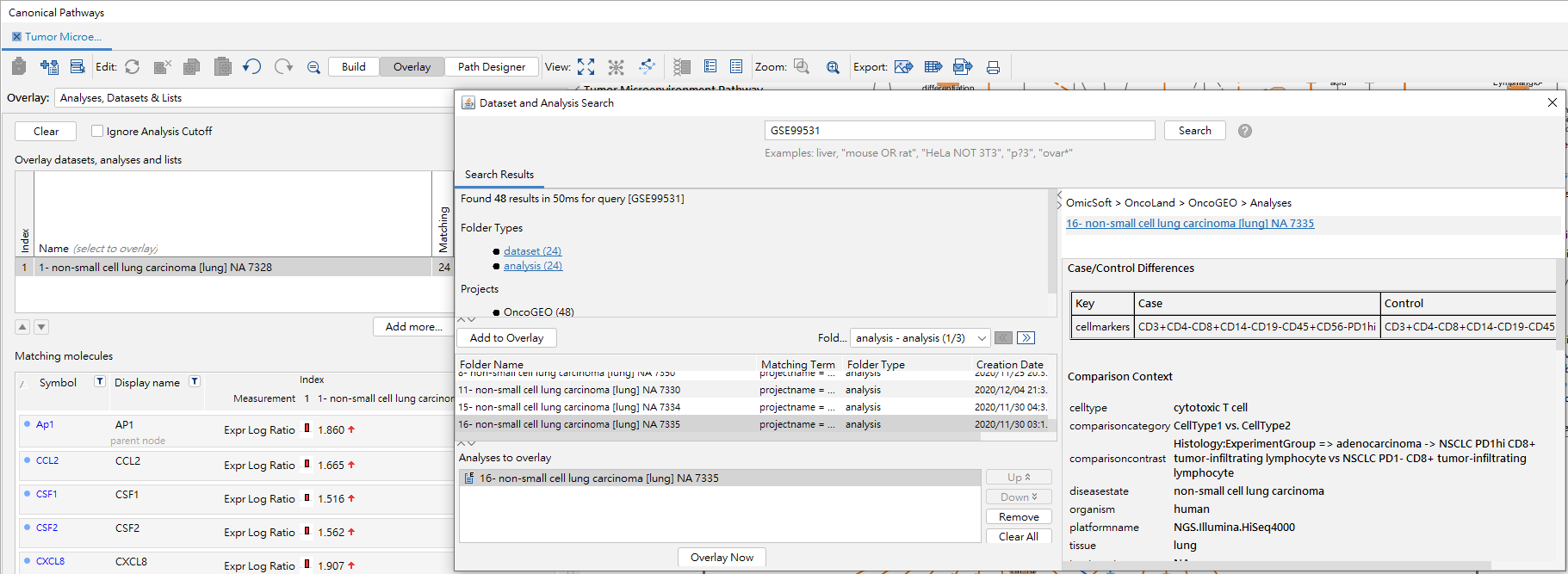
接著我們運用其他組肺癌的實驗資料,來比較TMP路徑中分子的變化,在Overlay -> Analyses, Datasets & Lists -> 選擇Add more,於搜尋列填上”GSE99531”後,可得到24組肺癌實驗數據及分析數據,這邊先選擇組別16後,點選Overlay Now以進行比較。
Overlay後可進一步比較兩組PD1於肺癌中表現不同的實驗資料,Ap1於組別1之Log Ratio為1.86,於組別16為-1.935,不同的活性表性,也進而影響了下游功能不同的表現。
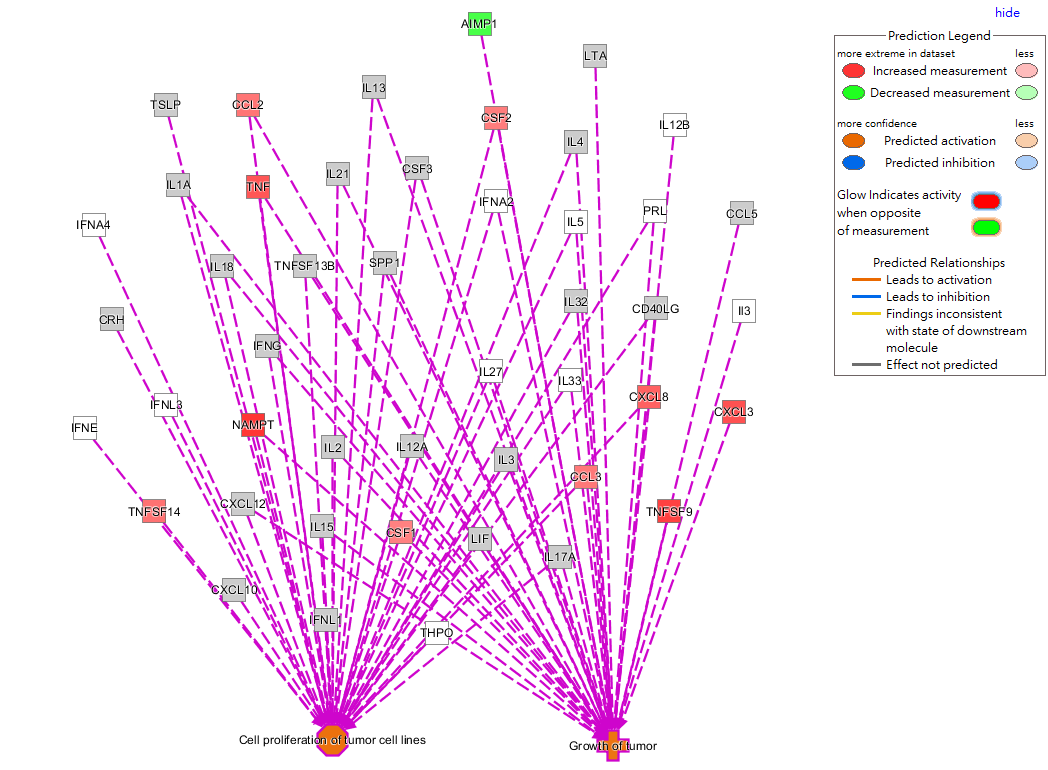
最後我們可以在回去IPA分析的頁面,觀看組別1的Upstream Analysis的分析,先篩選分子型態”Cytokine”,並將Z-score做排序,可得到71個活化非小細胞癌的上游調控因子,去探討文獻資料及實驗資料預測後,與腫瘤功能相關的調控機制。
上圖即為統整與Cytokine相關之上游調控因子,並加入腫瘤細胞增生(Cell proliferation of tumor cell lines)、腫瘤生長(Growth of tumor)功能之分子交互作用網路,可藉由實驗數據及IPA自動預測功能,快速得到分子與功能間上下游的關係,協助我們了解三者間的調控機制。
總結一下此次的範例介紹包含了基礎及進階功能的應用,我們可先透過IPA的資料庫建立,先探討有興趣的分子、疾病及路徑,並利用”connect”、”Path Explore”、”MAP”等功能串接IL1B, LC & TMP三者間關鍵的交互作用。
-
如何利用IPA資料庫搜尋,快速搜尋各分子之資料,及兩兩之間關係,並快速整理現有文獻資訊。
-
利用關鍵目標分子建立交互作用網路,並透過計算預測方式了解各分子間,是如何去影響網路內的成員。
再使用”Activity Plot”、”Core Analysis”、”Canonical Pathway”、”Upstream Analysis”等功能串接IL1B, LC & TMP三者,與實驗數據比對、路徑比較及上游調控預測。
-
使用真實實驗數據,探討腫瘤微環境路徑之表現變化及觀察不同數據集之間於同一路徑下之相似/不同變化。
-
分析真實實驗數據集去建立與免疫相關分子(如: 細胞激素),並探討中間調控之生物功能。
更多詳細資料可參考: Link
生圖購買的IPA是同時上線2人,因此須採取預約時段使用。申請註冊及預約說明請詳見IPA網頁。歡迎多多利用。
延伸閱讀:
如何利用QIAGEN IPA來研究腫瘤的微環境 (上)
利用IPA整合小鼠心肌新生之轉錄體學、蛋白質體學、代謝體學變化
IPA與冠狀病毒的應用
IPA 資料庫使用須知
Ingenuity Pathway Analysis (IPA) 資料庫介紹