本文經訊聯基因數位股份有限公司(原名:創源生技)同意轉載,原文出處:https://www.gga.asia/tc/Product_Information/BioInformatic/about_news_detail/4110
Reactome的Human Pathways現已收錄在IPA
二十年來,Reactome組織(reactome.org)一直在建立一個開源、開放查詢、人工校對並經過同儕審視的路徑資料庫,涵蓋了許多物種和主題領域。該組織已經導入與QIAGEN IPA相容的格式,並釋出他們的人類路徑資料庫。在此版本中,IPA已經完全整合了這502條路徑,提供更完整的資料查詢與分析服務。
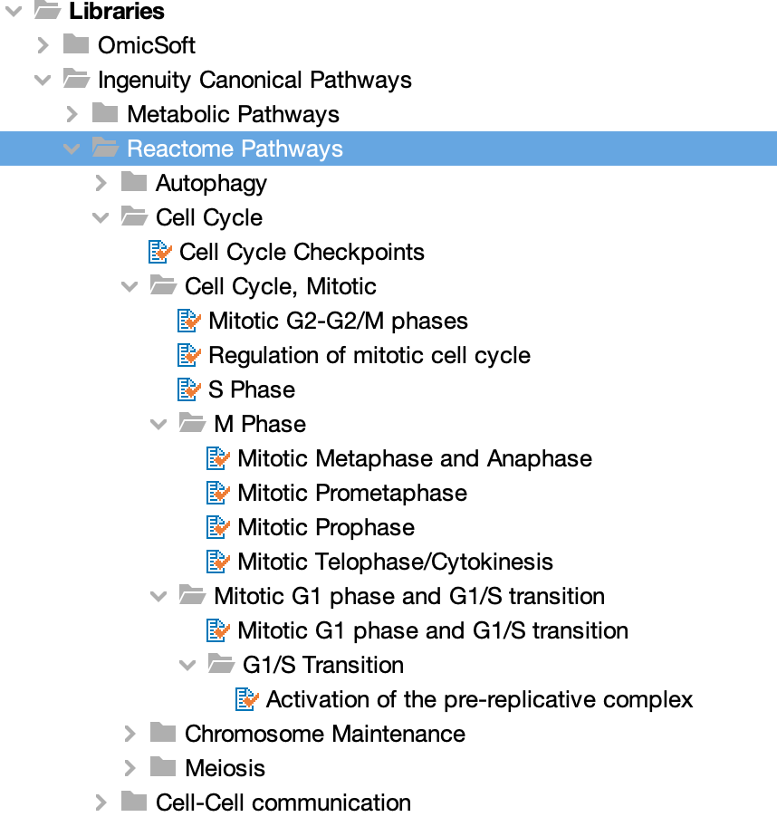
下圖1展示了IPA的Canonical Pathway的一部分,顯示了一些新的Reactome路徑的名稱和類別。
Reactome路徑在IPA的所有領域中都可用,包括Core Analysis、Build和Overlay tools,以及Search。以下使用了一個內胚層細胞的表達資料集來進行核心分析,它是來自人類誘導多能幹細胞(GSE66282)分化而來的,用來說明Reactome資料庫如何讓資料註解更豐富。圖2中突顯了加入了Reactome路徑,也更進一步強化IPA分析的準確度與豐富度。其中「最終內胚層的形成」路徑是所有路徑中得分最高的,而這預測結果與這資料集的本身的生物性質是相符合的。
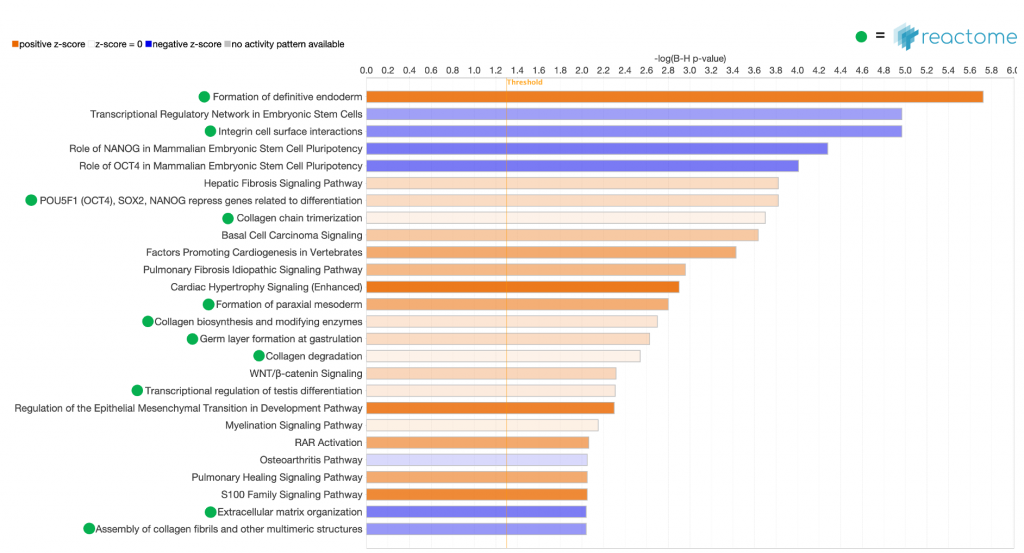
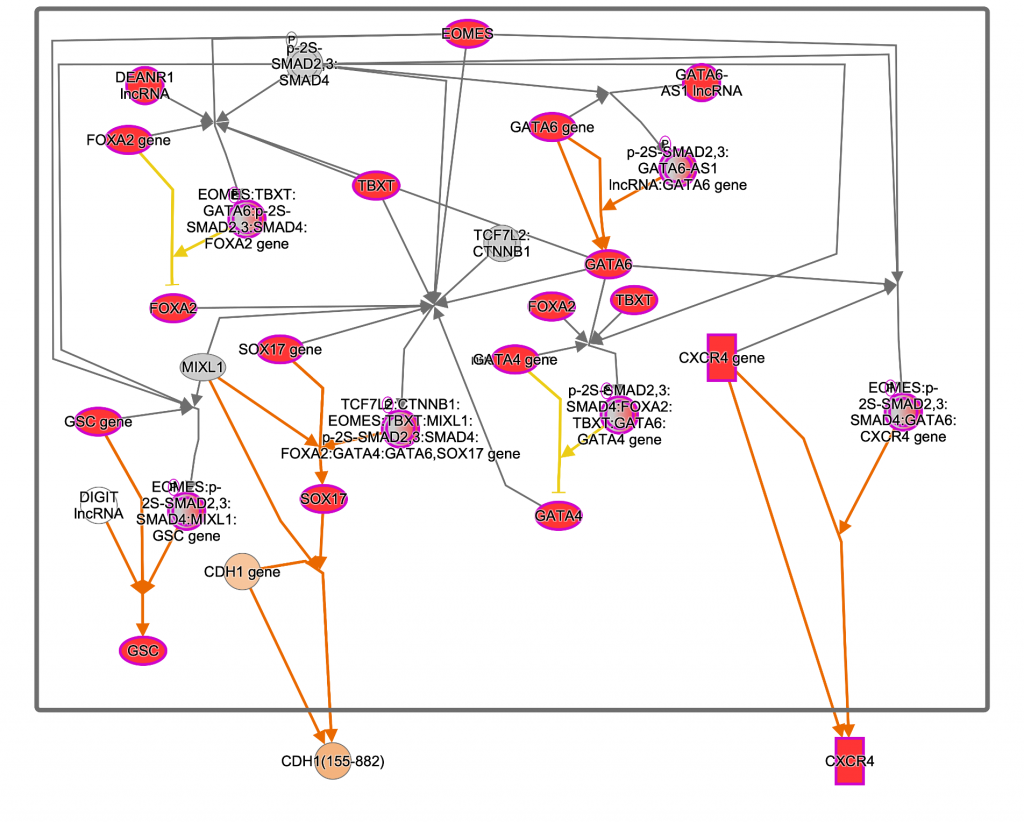
如圖3所示,IPA收錄了Reactome資料後,並以IPA內建的方式重新繪製並呈現。下圖是一些最終內胚層的生物標記,例如SOX17、GSC和FOXA2(如https://pubmed.ncbi.nlm.nih.gov/22236333所述)也如預期般正向調控的。
Reactome路徑包含大約10,300個基因。其中,大約5,600個基因與我們現有的Canonical pathways(本身包含8,700個基因)重疊。這意味著大約4,600個Reactome基因是我們Canonical pathways收藏的“新”基因。雖然來自Reactome的幾乎所有“新”基因已經與IPA中現有的疾病和功能相關聯,但總體效果是通過增強基因覆蓋範圍來改善IPA的Canonical pathways內容。
加速「網路連結關係相關文獻」的檢視方式
現在我們可以更快地查看每條連線的文獻證據了。雙擊任何一條關係連線,右側馬上就跳出相關的研究文獻,而無需加等待並彈出來另一個獨立視窗,大大加速查找的過程。
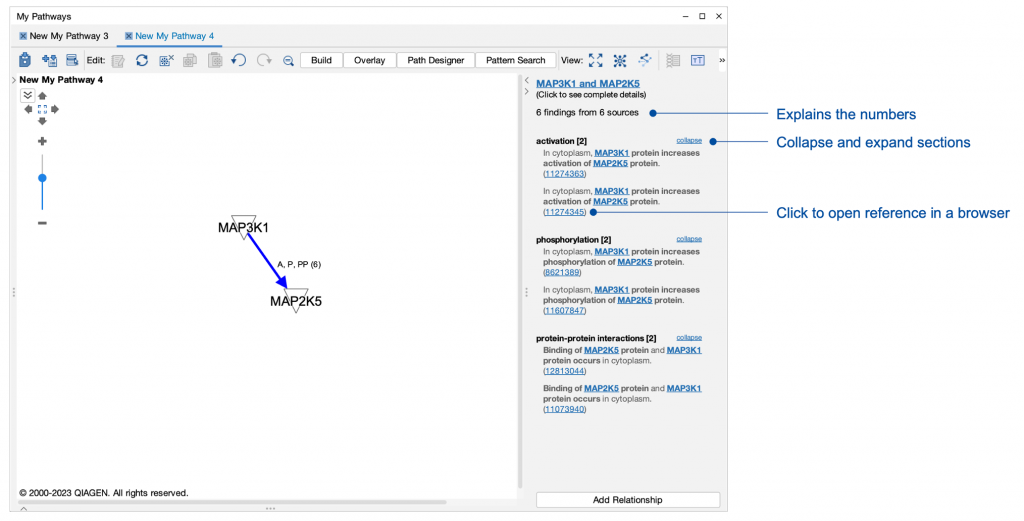
圖4. 新的快速關係查看側欄。現在查看結果不僅更快,還可以在面板中看到所有的結果,而無需加載單獨的結果頁面。
儲存Canonical pathways前次瀏覽設定
在核心分析中對Canonical pathways圖表所做的任何自訂都將與分析一併儲存。這將為您節省時間,尤其是當您進行大量自訂時,例如:為論文製作圖表時。
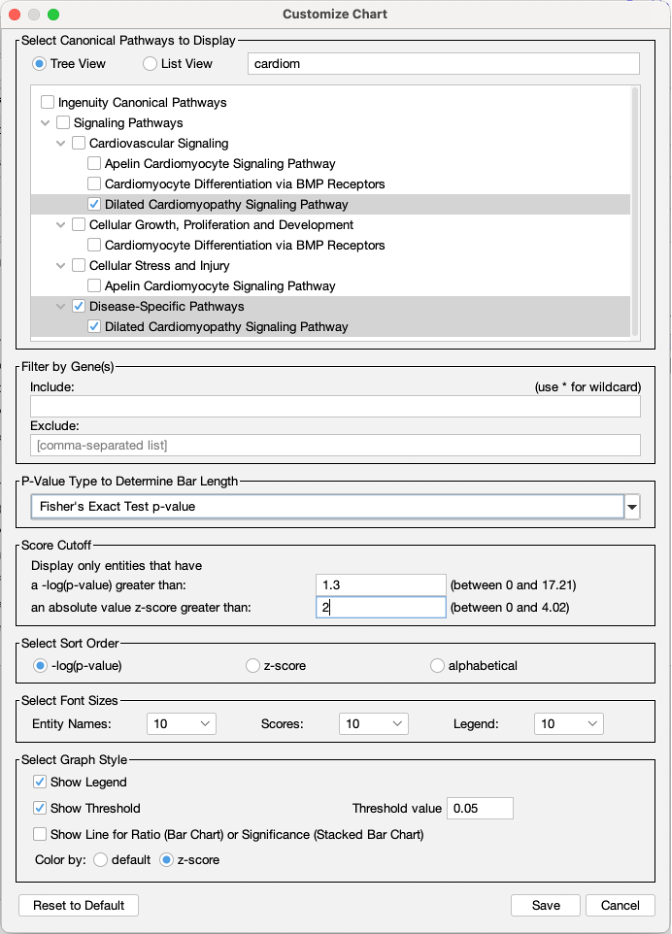
新的預設Canonical pathways圖表,並且會記住使用者上次瀏覽的類型
當改變預設是垂直呈現的Canonical pathways圖表類型成為水平條形圖後,之後您開啟一項分析時,將會預設為水平視圖。然而,IPA也會自動記住您最後查看的任何做過的核心分析的Canonical pathways圖表類型。例如,如果您打開其中一個分析並切換它以顯示Canonical pathways氣泡圖,然後關閉它,當您在未來返回查看時,它會記住以氣泡圖顯示Canonical pathways。
在IPA基因視圖的Land Explorer部分中的蛋白質組資料
Land Explorer現已擁有蛋白質組資料,該資料已與基因視圖連結。圖6展示了一個名為“蛋白質表達”的新部分。

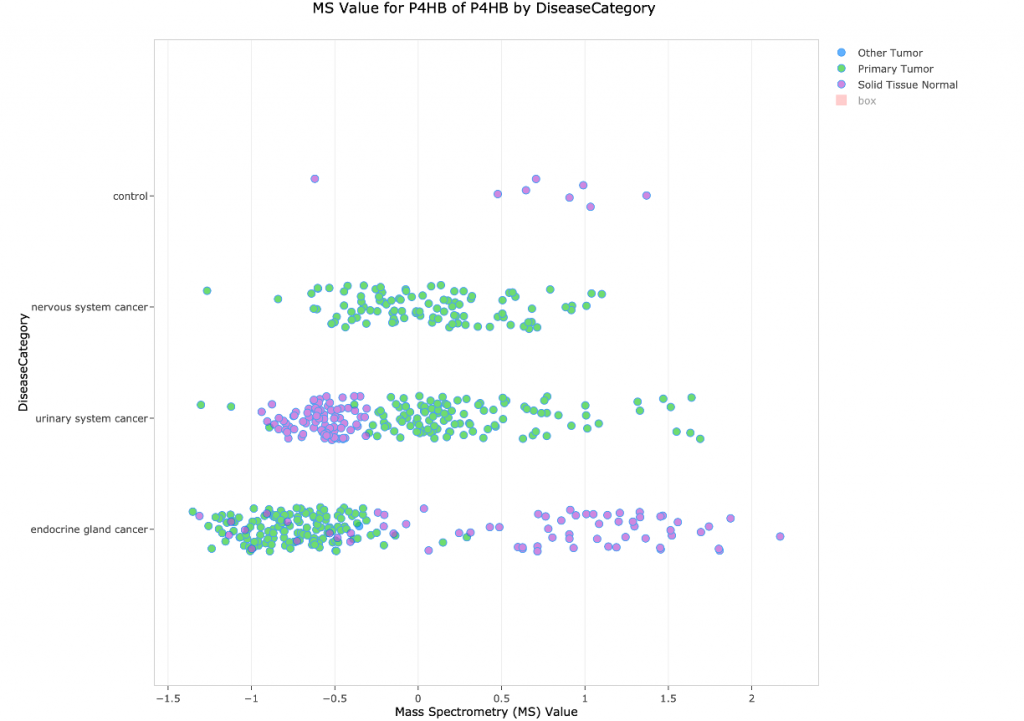
蛋白質體學是Land Explorer的一種新型資料,也適用於Analysis Match。例如,點選P4HB的基因視圖,並在蛋白質表達部分點擊一般腫瘤學鏈接,將在Land Explorer中顯示一個圖表(圖7),顯示其在新的Clinical Proteomic Tumor Land中編碼蛋白的質譜豐度值。這個地區的資料已經從Clinical Proteomics Tumor Analysis Consortium (CPTAC: https://proteomics.cancer.gov/programs/cptac)提供的資料中重新整理。
該圖表顯示由P4HB編碼的質酮醇4-羥基酶亞單位β蛋白的豐度(MS值)。與來自那些解剖區域的正常組織相比,該蛋白在內分泌腺瘤中的表達減少,而在泌尿系統癌症中的表達增加。
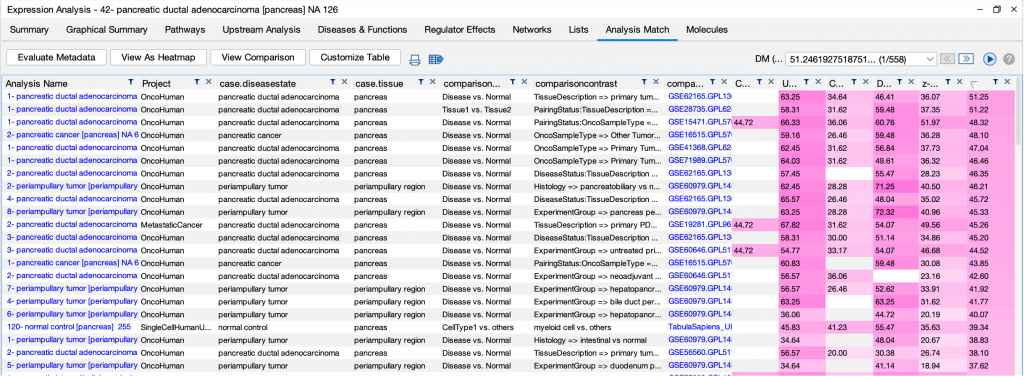
此外,CPTAC (https://proteomics.cancer.gov/programs/cptac) 提供了127個基於蛋白質的比較,這些比較已在IPA中分析並包含在Analysis Match中。圖8展示了對這些蛋白質組資料集的幾項分析的比較,突出了Canonical Pathway活化z分數的一些相似性和差異。
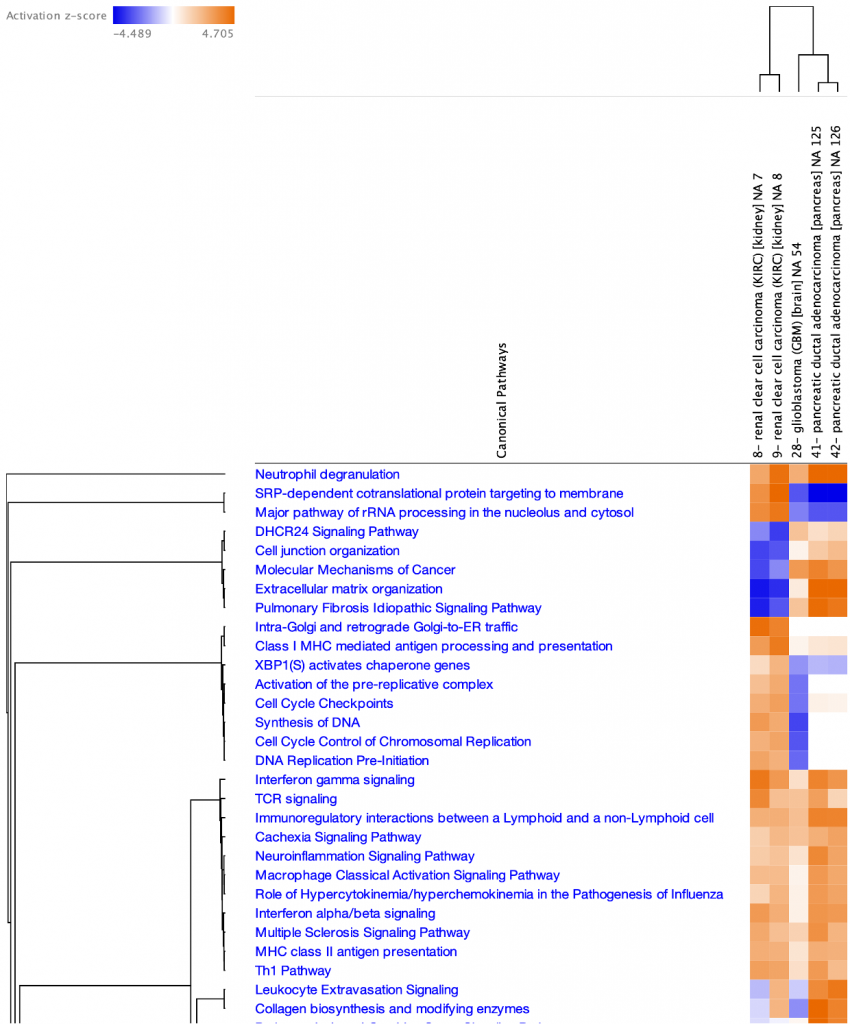
圖8. 幾個臨床蛋白質組資料集的典型路徑活化分數比較。胰腺導管腺癌分析結果聚集在一起,就像腎臟透明細胞癌分析一樣。
即使這些是差異蛋白質豐度的分析,它們也與圖9中顯示的來自RNA表達資料的分析相匹配,這是對於其中一項胰腺導管腺癌分析的展示。
圖9. 對於其中一個胰腺導管腺癌臨床蛋白質組資料集的分析匹配結果。主要腫瘤與正常相鄰組織的分析與許多其他來源的胰腺導管腺癌RNA表達分析相匹配,突顯了蛋白質和RNA水平表達之間的一致性。
在快速啟動視窗中的新手入門部分
一個新的「Getting started」標籤已經被添加到快速開始選單中,以幫助新用戶更快地熟悉IPA。您將在此處找到相關Help指示文件的連結。

Content updates 內容更新
6個全新路徑
- Autism Signaling Pathway自閉症訊號傳導路徑
- CGAS-STING Signaling Pathway
- NAFLD Signaling Pathway
- Pancreatic Secretion Signaling Pathway胰臟分泌訊號傳導路徑
- Protein Sorting Signaling Pathway蛋白質分類訊號傳導路徑
- ROBO SLIT Signaling Pathway
5個現有的路徑已更新,以包含活動模式
- Hepatic Cholestasis 肝性膽鹽滯留
- Docosahexaenoic Acid (DHA) Signaling二十二碳六烯酸(DHA)訊號傳導
- FXR/RXR Activation
- Molecular Mechanisms of Cancer癌症的分子機制
- Netrin Signaling Netrin訊號傳導
新增超過350,000項新發現(使IPA的總收錄數超過1,299萬項)
- >118,000 Expert findings (from literature curation)
- >127,000 Cancer mutation findings from ClinVar
- >10,800 Protein–protein interaction findings from BioGrid
- >3,900 Protein–protein interaction findings from IntAct
- >1,300 Target-to-disease findings from ClinicalTrials.gov
- >1,300 Drug-to-disease findings from ClinicalTrials.gov
- >600 gene-to-cancer findings from the Catalogue of Somatic Mutations in Cancer (COSMIC)
- >350 Gene Ontology findings
- >50 findings from the Human Metabolome Database (HMDB)
- >20 findings from the Mouse Genome Database (MGD)
- >200 newly mappable chemicals
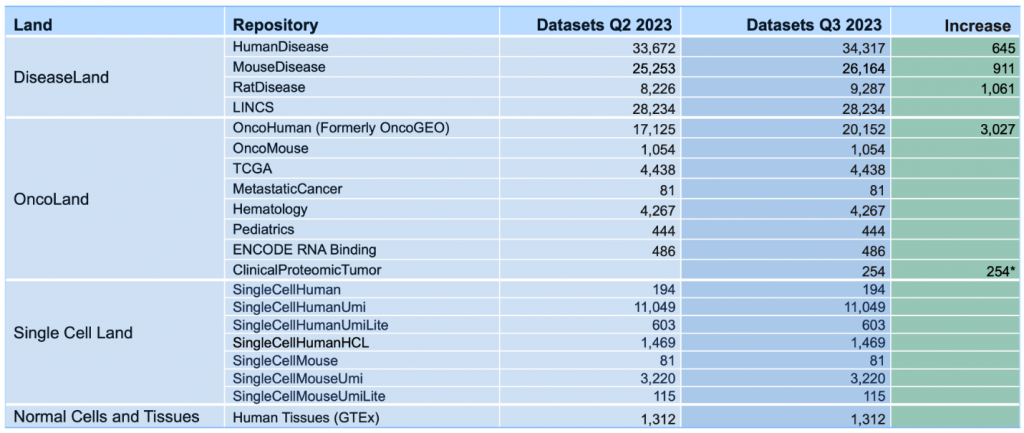
147,221個表現資料集(新增5,898個)
這次的OmicSoft分析版本首次包含蛋白質組資料。來自CPTAC (https://proteomics.cancer.gov/programs/cptac) 的127個基於蛋白質的比較。這些將出現在IPA的一個新Land中,名為ClinicalProteomicsTumor。