如果我們有一條序列,想知道上面有沒有miRNA targets,有什麼工具可以幫我們完成呢?
今天就來介紹一個歷史悠久,預測miRNA targets的好工具-miRanda。
miRanda的演算法發表自2003年,也是全世界第一個發表miRNA target的預測軟體,跟其他相似的分析工具(如TargetScan, PicTar)比起來,miRanda不但提供了搜尋網頁(http://www.microrna.org/),還提供軟體下載,甚至開放了軟體原始碼可以讓使用者自行修改程式,是不是很棒呢?
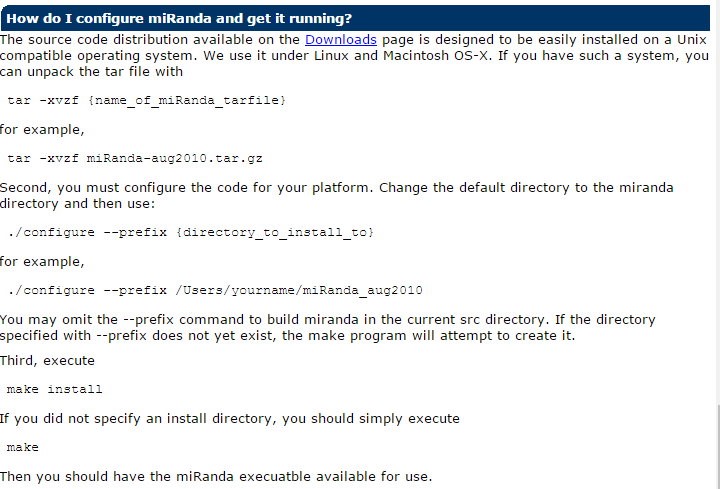
讓我們先下載軟體來使用吧,請注意:miRanda必須在Linux或是Macintosh OS-X環境下執行。
首先連到miRanda網站(http://www.microrna.org/),然後點Download
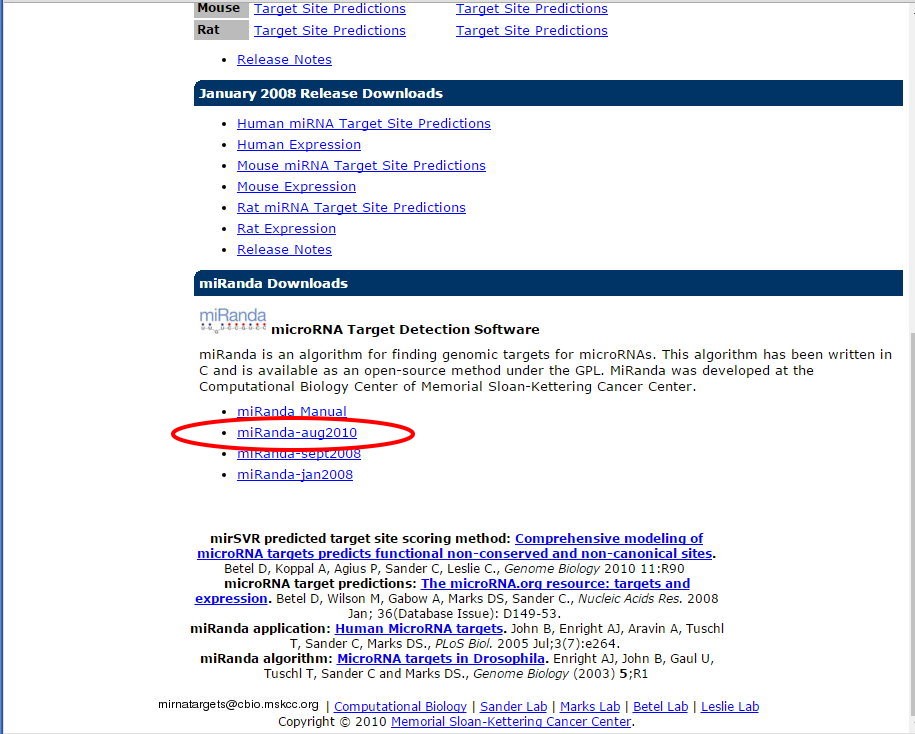
將頁面往下捲,選擇miRanda-aug2010下載
下載後需透過指令模式才能使用,例如Mac OS-X的應用程式”終端機”。
將下載的檔案解壓縮並安裝,步驟可參考檔案INSTALL或是網站上FAQ(如下圖)
接下來就可以直接執行了 (使用手冊 http://cbio.mskcc.org/microrna_data/manual.html)
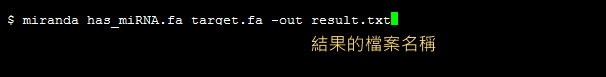
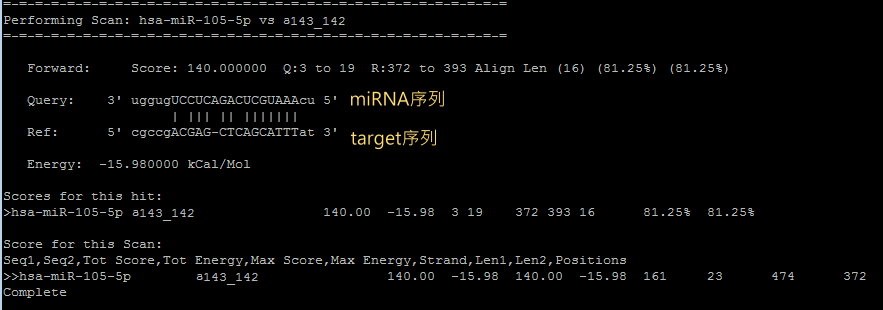
將miRNA序列和你的序列分別存成FASTA格式的文字檔,比如我把所有human miRNAs序列存成has_miRNA.fa,要找target的序列存成target.fa,基本指令如下:
結果如下:
每一筆預測結果都會計算分數(Score),每個鹼基配對(base-pairing)會得到一個特定分數:
A:U = 5
G:C = 5
G:U = 2
Gap opening: -9.0
Gap extension -4.0
All other base pairs (mismatches) = -3
另外每一個鹼基配對位置s(i)會乘上一個權重值w(i),位置2-8屬於seed區域,權重值較高。
Position s(i) |
Weight w(i) |
1 |
1.0 |
2-8 |
4.0 |
9-21 |
1.0 |
最後將所有位置的數值加總,就是miRNA target的分數(Score)
Score = SUM(over i) [ w(i) s(i) ]
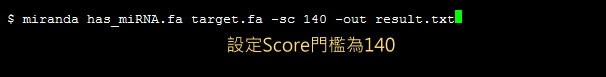
我們也可以設定Score門檻,比如Score必須大於140,這樣可以得到較準確的預測值
如果覺得得到的結果仍然太多,miRanda提供很多更嚴格的設定條件(如energy,自訂penalty數值,不允許任何mismatch出現在seed region等),請參考使用者手冊 (http://cbio.mskcc.org/microrna_data/manual.html)
謝謝分生所Dr.徐唯哲提供